Abstrak
Lakase termasuk dalam superfamili multicopper oxidases (MCO), sekelompok enzim dengan kemampuan untuk mereduksi oksigen ke air dalam suatu reaksi tanpa menghasilkan produk sampingan yang berbahaya. Aktivitas lakase dipengaruhi oleh banyak faktor, seperti struktur; jumlah, lokasi, dan status pengikatan ion tembaga; dan status pengikatan substrat. Sejumlah besar sekuens yang belum dikarakterisasi secara eksperimental telah dianotasi sebagai lakase. Namun, fungsi biologis MCO yang dikarakterisasi dianggap bervariasi, dan spektrum substrat tumpang tindih dengan MCO lainnya. Di sini, kami mengidentifikasi 34 putative bacterial laccase sequences dari data metagenom untuk air limbah industri. Kami menggunakan alat pembelajaran mesin untuk menyaring enzim dengan aktivitas lakase dengan menggabungkan kapasitas pengikatan tembaga T1, kapasitas pengikatan tembaga keseluruhan, dan kapasitas pengikatan substrat. Kami juga menggunakan perbandingan perangkat lunak untuk menghilangkan sekuens dengan perbedaan besar antara aplikasi perangkat lunak yang berbeda. Struktur tiga dimensi enzim yang diidentifikasi diprediksi menggunakan alphafold , posisi ion logam dalam protein diprediksi oleh metal3d dan autodock-vina , dan docking mereka dengan ABTS [yaitu 2,2′-azinobis(3‑ethylbenzo-6‑thiazolinesulfonic acid)] sebagai substrat diprediksi oleh rosetta dan autodock-vina . Berdasarkan hasil docking, kami memilih 10 protein dengan skor tinggi, dua protein dengan skor rendah dan satu protein komposit untuk ekspresi menggunakan vektor pET-21d (+). Sejalan dengan prediksi kami, semua protein dengan skor tinggi yang dipilih menunjukkan aktivitas terhadap ABTS. Secara keseluruhan, kami menjelaskan metode untuk menemukan dan merancang oksidase multitembaga seperti lakase bakteri yang baru, yang menawarkan kemungkinan yang lebih besar untuk degradasi berbagai komponen berbahaya yang berasal dari polusi lingkungan.
Singkatan
Bahasa Indonesia: ABTS
2,2′-azinobis(asam 3-etilbenzo-6-tiazolinsulfonat)
DUF152
domain fungsi yang tidak diketahui 152
Bahasa Indonesia: IPTG
isopropil tio-β- d -galaktosida
LMCO
oksidase multitembaga mirip lakase
MCO
oksidase multitembaga
Ditemukan pada tahun 1883, lakase termasuk dalam superfamili multitembaga oksidase (MCO), sekelompok enzim dengan fungsi biologis beragam dan kemampuan untuk mereduksi oksigen menjadi air dalam suatu reaksi tanpa menghasilkan produk sampingan yang berbahaya [ [ 1 – 3 ] ]. Misalnya, poli-tembaga oksidase dan lakase telah ditemukan pada jamur, tumbuhan tingkat tinggi, bakteri, dan serangga. Mereka terlibat dalam biodegradasi lignin, produksi pigmen, dan patogenesis tumbuhan. Pada tumbuhan, mereka memiliki fungsi potensial dalam biosintesis lignin [ [ 2 ] ]. Sebagian besar lakase yang telah dipelajari dan digunakan dalam proses industri berasal dari jamur dan umumnya berukuran 60–100 kDa. Lakase dari eukariota cenderung mengalami glikosilasi [ [ 4 ] ]. Mereka menggunakan oksigen molekuler untuk mengoksidasi berbagai macam senyawa aromatik dan non-aromatik melalui mekanisme reaksi yang dikatalisis oleh radikal bebas, yang sering kali terlibat dalam proses degradasi lignin [ [ 2 , 5 ] ]. MCO dengan aktivitas lakase pada prokariota sering kali digambarkan sebagai ‘polifenol oksidase’, ‘multi-tembaga oksidase’ atau ‘enzim seperti lakase’. Enzim-enzim dalam famili MCO memiliki struktur molekuler yang serupa dan juga fungsi katalitik yang serupa dan profil substrat yang tumpang tindih. Mereka dibagi menjadi empat kelompok penting: lakase ( EC1.10.3.2 ), askorbat oksidase ( EC1.10.3.3 ), ferroksidase ( EC1.16.3.1 ) dan nitrit reduktase ( EC1.7.2.1 ) [ [ 6 ] ]. Mereka semua mengkatalisis oksidasi substrat lakase yang khas. MCO bakteri dengan aktivitas lakase selalu memiliki banyak fungsi [ [ 7 ] ]. Sebagian besar lakase bakteri yang diekspresikan secara alami berada di dalam sel [ [ 8 ] ]. Lebih jauh lagi, sebagian besar lakase bakteri yang teridentifikasi belum dilaporkan terlibat secara langsung dalam degradasi lignin [ [ 9 , 10 ] ]. Pada bakteri, mereka dilaporkan berperan dalam melanogenesis, resistensi cangkang spora, morfogenesis dan detoksifikasi tembaga [ [ 11 , 12 ] ]. Suhu reaksi optimal untuk sebagian besar enzim lakase jamur berkisar antara 25 hingga 50 °C [ [ 1 , 13 ] ]; namun, lakase bakteri biasanya memiliki suhu reaksi optimal yang relatif tinggi, sehingga meningkatkan luasnya aplikasi lakase [ [ 14 ] ].
Urutan asam amino lakase menunjukkan identitas rendah satu sama lain, tetapi struktur tiga dimensinya serupa [ [ 5 ] ]. Akseptor elektron awal dalam reaksi redoks yang dikatalisis oleh lakase adalah ion tembaga T1, yang terletak di kantong yang dekat dengan permukaan enzim. Lakase bakteri memiliki potensi redoks yang rendah, sedangkan lakase dengan potensi redoks tinggi banyak ditemukan pada jamur [ [ 9 , 10 ] ]. Reduksi ion tembaga T1 adalah langkah pembatas laju dalam reaksi yang dikatalisis lakase, dan tempat pengikatan ion tembaga T1 juga menjadi fokus penyaringan dan desain lakase [ [ 15 , 16 ] ]. Senyawa fenolik dioksidasi oleh lakase menjadi radikal fenoksi. Namun, reaksinya reversibel, berdasarkan stabilitas radikal fenoksi [ [ 17 ] ]. Daur ulang redoks substrat fenolik sebagai mediator memperluas jangkauan substrat lakase [ [ 18 , 19 ] ]. Kapasitas oksidatif non-spesifik yang tinggi dari lakase dan fakta bahwa oksigen molekuler bertindak sebagai akseptor elektron menjadikan mereka kandidat yang sangat baik untuk aplikasi industri dan biologi [ [ 20 ] ]. Lakase memiliki banyak aplikasi potensial, seperti dalam kimia organik, produksi biofuel dan bioremediasi, serta dalam industri makanan, kertas, tekstil, furnitur, konstruksi, cat, kosmetik dan biomedis [ [ 21 – 23 ] ].
Lakase tipikal memiliki empat ligan tembaga dan massa molekul dalam kisaran 50–70 kDa atau lebih besar, sedangkan lakase atipikal memiliki massa molekul antara 20 dan 40 kDa, dengan seng lebih sering menggantikan ion tembaga [ [ 15 ] ]. Keluarga enzim lakase domain of unknown function 152 (DUF152), seperti RL5, BT4389 dan YfiH, menunjukkan aktivitas dalam oksidasi senyawa aromatik. RL5 telah ditunjukkan oleh spektrometri massa masih mengandung empat atom tembaga per molekul enzim [ [ 24 , 25 ] ]. Massa molekul dari 11 lakase aktif yang diperoleh dalam penelitian ini berkisar antara 24 hingga 28,5 kDa. Sejumlah besar sekuens yang belum dikarakterisasi secara eksperimental telah dianotasi sebagai lakase. Namun, fungsi biologis dari MCO yang dikarakterisasi diasumsikan bervariasi, dan spektrum substrat tumpang tindih dengan MCO lainnya [ [ 6 ] ]. Enzim dalam penelitian ini disebut laccase-like multicopper oxidase (LMCO).
Sebagai hasil dari kemajuan dalam teknologi informasi dan sekuensing, metagenomik telah menghasilkan sejumlah besar data sekuensing, sehingga memungkinkan kita untuk mendapatkan sekilas potensi mikroorganisme yang tidak dikultur dan mencari LMCO baru [ [ 26 , 27 ] ]. Namun, akurasi anotasi fungsional tradisional perlu ditingkatkan, dan identifikasi eksperimental memakan waktu dan padat karya. Pemodelan dan analisis sekuens protein melalui teknologi informasi baru dan pembelajaran mesin akan membuka jalan ke depan. Empat teknologi, yaitu alphafold , metal3d , rosetta dan autodock-vina , mewakili alat yang sangat baik dalam pencarian LMCO baru. alphafold memungkinkan prediksi struktur protein dengan presisi atomik, bahkan ketika struktur serupa tidak diketahui [ [ 28 , 29 ] ]. Menggunakan algoritma pembelajaran mendalam, kesalahan alphafold 2 kurang dari lebar atom [ [ 30 ] ]. metal3d memprediksi lokasi ion logam dalam protein dan mengeluarkan ukuran kepercayaan untuk setiap situs yang diprediksi; ini juga berlaku untuk protein dengan beberapa ortolog dalam basis data protein [ [ 31 ] ]. autodock-vina dengan cepat memungkinkan docking molekuler dan penyaringan virtual pada CPU multicore [ [ 32 ] ]. Selain itu, sebagai perangkat lunak awal untuk memprediksi interaksi protein-molekul kecil, rosetta Ligand telah ditingkatkan sejak 2006 untuk memungkinkan fleksibilitas dalam tulang punggung reseptor dan fleksibilitas yang lebih besar dalam ligan, dan rosetta-highresdocker telah lebih jauh meningkatkan fleksibilitas penggunaan [ [ 33 , 34 ] ]. Interface_delta_X dalam keluaran rosetta mewakili perbedaan antara kompleks protein-ligan yang terikat dan protein dan ligan yang tidak terikat dengan Rosetta Energy Units. Alat-alat ini telah sangat meningkatkan akurasi dan efisiensi penemuan LMCO baru.
Dalam studi ini, (a) urutan LMCO yang berpotensi hadir dalam air limbah industri dicari, menggunakan data sekuensing metagenom sebagai titik awal. (b) Prediksi struktur tiga dimensi LMCO potensial dilakukan menggunakan alphafold 2. (c) Prediksi posisi ligan logam dengan metrik keyakinan atau afinitas dibuat menggunakan metal3d dan autodock-vina . (d) Prediksi afinitas protein terhadap substrat seperti 2,2′-azinobis(3‑ethylbenzo-6‑thiazolinesulfonic acid) (ABTS) dibuat menggunakan rosetta-highresdocker dan autodock-vina . (e) Menggunakan metode simulasi di atas, fragmen yang cocok dari LMCO lain yang ada dicari untuk melengkapi urutan LMCO yang tidak lengkap. (f) Dalam pengukuran aktivitas LMCO, 11 LMCO aktif ditentukan, dan parameter dan metode untuk menyaring LMCO dengan prediksi dan analisis struktur divalidasi. Studi ini memanfaatkan pengembangan teknologi pengurutan dan informasi untuk menyaring dan merancang LMCO baru yang memenuhi kebutuhan yang ditentukan.
Bahan dan metode
Bahan kimia dan reagen
Total DNA mikroorganisme diisolasi dari cekungan air limbah Neot-Hovav di Israel, yang berisi limbah industri. Air limbah dari cekungan ini dicirikan oleh konsentrasi garam yang tinggi lebih dari 160 g·L −1 dan keberadaan sejumlah besar senyawa organik terhalogenasi yang beracun [ [ 35 ] ].
Phusion High-Fidelity PCR Master Mix, FastDigest, T4 DNA Ligase, fragmen DNA, primer oligonukleotida, B-PER Complete Bacterial Protein Extraction Reagent, Qubit dsDNA Assay Kits dan Ni-NTA Spin Columns dibeli dari Thermo Fisher Scientific (Waltham, MA, AS). (ABTS), fenol/kloroform/alkohol isoamyl dan isopropil tio-β- d -galaktosida (IPTG) dibeli dari Merck (Darmstadt, Jerman).
Pengurutan metagenom
Pustaka sekuensing Illumina disiapkan menggunakan Illumina Nextera DNA Sample Preparation Kit dan Nextera Index Kit untuk multiplexing sebagaimana direkomendasikan oleh produsen (Illumina Inc., San Diego, CA, AS). Untuk menilai kualitas dan ukuran pustaka, sampel dianalisis dengan Agilent Bioanalyzer 2100 menggunakan Agilent High-Sensitivity DNA Kit sebagaimana direkomendasikan oleh produsen (Agilent Technologies, Waldbronn, Jerman). Konsentrasi pustaka ditentukan menggunakan Qubit ® dsDNA HS Assay Kit sesuai dengan petunjuk produsen (Life Technologies GmbH, Darmstadt, Jerman). Sekuensing dilakukan menggunakan instrumen HiSeq2500 (Illumina Inc.), menggunakan HiSeq ® Rapid SBS Kit v2 (500 Cycle) dan HiSeq ® Rapid PE Cluster Kit v2 untuk sekuensing dalam mode paired-end dengan 2 × 250 siklus. Hasil pembacaan mentah disaring kualitasnya menggunakan trimmomatic v0.39 (x1) dan selanjutnya dirakit secara de novo dengan metaspades v3.11.1 (x2). Kontig yang lebih besar dari 1 kb diberi anotasi dengan prokka 1.12.0 (x3) dalam mode meta untuk meningkatkan prediksi gen untuk genom yang sangat terfragmentasi [ [ 36 – 38 ] ].
Prediksi struktur tiga dimensi
Prediksi struktur tiga dimensi dilakukan menggunakan NVIDIA Tesla T4 ( https://www.nvidia.com/en-gb/datacenter/teslat4 ) yang menjalankan alphafold 2 untuk 34 kemungkinan urutan LMCO yang diberikan oleh hasil sekuensing. Untuk mencapai akurasi terbaik, semua prediksi bekerja dengan parameter ‘-model_preset=mono_casp14’.
Prediksi posisi ligan logam
Prediksi posisi ligan logam dilakukan menggunakan metal3d dan autodock-vina . metal3d dijalankan dengan parameter berikut: ‘–metalbinding –writeprobes –softexit’. Empat ion tembaga pertama dalam hasil dengan daerah pengikatan yang jelas berbeda dan metrik keyakinannya dikumpulkan. autodock-vina pertama kali digunakan untuk melakukan docking global protein dengan ion tembaga. Setelah itu, dengan empat posisi ligan logam dengan skor terbaik dalam hasil yang diprediksi oleh metal3d , autodock-vina dijalankan lagi untuk docking lokal yang berpusat pada masing-masing dari mereka. Hasil afinitas dikumpulkan, menyaring hasil berdasarkan di mana autodock-vina tidak bertentangan dengan hasil metal3d untuk memilih model terbaik. Hasil metal3d , yang cocok dengan hasil autodock , digunakan; skor ion tembaga terbaik dicatat; dan rata-rata dari empat skor ion tembaga teratas dihitung.
Dermaga untuk substrat
Dengan menggunakan berkas ‘sdf’ dari ABTS (CID: 9570474) yang diunduh dari PubChem untuk menjalankan autodock-vina , nilai afinitas dihitung. Dengan menggunakan parameter ‘-nstruct 50 000’ yang berpusat pada tiga hasil pertama yang diberikan oleh autodock-vina , rosetta-highresdocker dijalankan untuk melakukan dock, dan nilai ‘interface_delta_X’ dihitung untuk memilih model terbaik.
Memperbaiki urutan LMCO yang tidak lengkap
Seperti yang ditunjukkan pada Gambar 1 , satu sekuens LMCO yang diprediksi, Lac6, ditemukan tidak memiliki sebagian besar sekuens N-terminalnya. Hasil prediksi dan docking menunjukkan bahwa situs pengikatan tembaga T1 lengkap yang khas (His…Cys…His) tidak dapat dibentuk. Untuk mengatasi hal ini, sekuens ‘HAGWRG’ dipilih untuk penyambungan berdasarkan konservasi situs pengikatan tembaga T1 dan kesamaan struktural di antara MCO. Setelah penyelarasan sekuens, sekuens N-terminal dari LMCO lain yang diprediksi digunakan untuk memperbaiki Lac6. Sekuens yang diperbaiki menjalani prediksi lebih lanjut dan analisis docking, dan hasil optimal dipilih. Fragmen DNA diperoleh menggunakan PCR fusi [ [ 39 ] ].
 Gbr. 1
Gbr. 1
Buka di penampil gambar
Kekuatan Gambar
Penyelarasan berganda dari sekuens lakase-seperti multitembaga oksidase (LMCO) yang diprediksi dalam penelitian ini menggunakan perangkat lunak geneious dengan ‘ clustal omega ‘ dan perangkat lunak genedoc untuk menggambar. Sekuens LMCO yang dilestarikan dalam penelitian ini diberi nama CS_Lac. Sekuens yang dilestarikan dari keluarga DUF152 dari NCBI (pfam02578) diberi nama Cs_DUF152 [ [ 46 ] ]. Residu merah dalam sekuens yang dilestarikan diperoleh menggunakan opsi Color Bits for Identity.
Ekspresi dan pemurnian
Urutan DNA dihasilkan menggunakan penggunaan kodon Escherichia coli dan alat NovoPro dan disintesis oleh Thermo Fisher Scientific. Fragmen DNA sintetis diligasikan ke dalam vektor pET-21d(+) menggunakan endonuklease restriksi Nco I dan Xho I dengan ligase DNA T4, dan sel E. coli BL21(DE3) ditransformasi dengan produk ligasi. Menggunakan medium LB dengan gliserol 5% dan ampisilin 100 μg·mL −1 , kultur diinkubasi hingga D 600 sebesar 0,7 ± 0,05 tercapai. IPTG pada konsentrasi akhir 1 mm dan CuSO4 pada konsentrasi akhir 0,25 mm kemudian ditambahkan, dan campuran diinkubasi pada suhu 37 °C selama 4 jam. Kultur dikumpulkan dan disentrifugasi selama 5 menit pada 1800 g , dan B-PER Complete Bacterial Protein Extraction Reagent dengan 5 mL reagen·g −1 biomassa ditambahkan untuk menangguhkan kembali dan melisiskan sel. Fraksi sel yang diperoleh digunakan untuk mengonfirmasi ekspresi yang berhasil dengan SDS/PAGE, serta untuk menentukan aktivitas enzim secara kualitatif. Media dan parameter yang sama digunakan untuk induksi semalam pada 16 °C, kultur dikumpulkan dan disentrifugasi selama 5 menit pada 1800 g , salin penyangga fosfat digunakan untuk menangguhkan kembali sel, dan ultrasound digunakan untuk mengganggu sel. Kolom Spin HisPur™ Ni-NTA digunakan untuk memurnikan protein target. Buffer elusi adalah salin penyangga fosfat dengan 250 m m imidazol. Konsentrasi protein ditentukan menggunakan Rotiquant (Carl ROTH, Karlsruhe, Jerman), sesuai dengan petunjuk pabrik.
Pengukuran aktivitas enzim
Fraksi seluler digunakan untuk mengukur aktivitas enzim secara kualitatif, dan kinetika LMCO diukur menggunakan protein yang dimurnikan. Reaksi enzimatik diuji menggunakan Spektrofotometer DS-11+ (DeNovix, Wilmington, DE, AS) pada 420 nm dengan konsentrasi akhir 100 mm buffer asetat (CH3COOH dengan CH3COONa , pH 4,6) dengan 2,5 mm tembaga sulfat, menggunakan 0,5 mm ABTS (PanReac AppliChem, Chicago, IL, AS; ε 420 = 38.000 m −1 ·cm −1 ) sebagai substrat. BL21 (DE3) yang mengandung pembawa plasmid kosong yang diinduksi oleh kultur dalam kondisi yang sama digunakan sebagai kontrol negatif saat mengukur aktivitas enzim secara kualitatif. Untuk pengukuran kuantitatif aktivitas enzim menggunakan protein yang dimurnikan, buffer elusi yang dimurnikan digunakan sebagai kontrol negatif. Aktivitas enzim diukur pada pH 3,8, 4,0, 4,2, 4,4, 4,6, 4,8, 5,0, 5,2, 5,4, 5,6 dan 5,8 dan pada suhu 37, 45, 50, 55, 60, 65, 70, 80 dan 90 °C menggunakan buffer pH optimal. Pengukuran kualitatif dilakukan pada suhu 37 °C dengan pH 4,6. Kinetika Michaelis–Menten diukur menggunakan ABTS pada konsentrasi akhir 10–200 μm .
Ion tembaga digantikan dengan ion seng untuk pengukuran aktivitas kualitatif selama inkubasi, induksi dan uji aktivitas enzim.
Hasil dan Pembahasan
Analisis urutan lakase
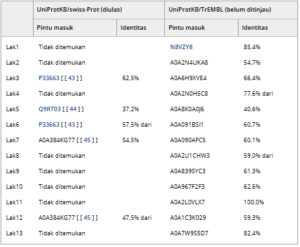
Kami mengidentifikasi 34 putative LMCO sequences dalam 179.233 predicted amino acid sequences yang diperoleh oleh prokka 1.12.0 (x3). Data ini berasal dari dataset awal yang berisi 48.119 genom yang telah dirakit. LMCO yang digunakan dalam penelitian ini diberi nama Lac1 hingga Lac13. Urutan ini mengalami blast dengan basis data UniProtKB/Swiss-Prot (yang telah diulas), yang diberi anotasi secara manual dan berisi hasil percobaan, dan basis data UniProtKB/TrEMBL (yang belum diulas) yang diberi anotasi komputer. Seperti yang ditunjukkan dalam Tabel 1 , yang menampilkan hasil BLAST, sebagian besar urutan tidak cocok dengan urutan dalam UniProtKB/Swiss-Prot (yang telah diulas), dan sebagian besar urutan dalam penelitian ini yang cocok dengan urutan dalam UniProtKB/TrEMBL (yang belum diulas) juga memiliki identitas di bawah 70%. Tak satu pun enzim yang ditinjau dan cocok telah dikarakterisasi untuk aktivitas lakase atau MCO. Semua sekuens yang ditinjau dan cocok diidentifikasi dan sekuens yang tidak ditinjau diberi anotasi sebagai fosforilase nukleosida purin. Telah ditunjukkan bahwa protein DUF152 (YfiH) memiliki potensi untuk mengkatalisis tidak hanya substrat lakase yang khas, tetapi juga analog nukleosida. Itulah sebabnya kelas protein ini juga sering diberi anotasi sebagai fosforilase nukleosida purin. DSM 5159 dari Thermomicrobium roseum memiliki aktivitas lakase dan fosforilase nukleosida purin [ [ 40 ] ]. PsLac dari Peribacillus simplex menggunakan fosforilase nukleosida purin sebagai templat untuk pemodelan homologi dan akhirnya memvalidasi aktivitas lakase [ [ 41 ] ]. Id17_09670 juga tidak menemukan kecocokan yang signifikan dalam basis data dan dicocokkan di Uniport dengan fosforilase nukleosida purin. Namun, diperkirakan bahwa senyawa ini kemungkinan besar memiliki aktivitas lakase [ [ 42 ] ].
Tabel 1. Hasil BLAST Uniprot. Lac1 – Lac13 merupakan sekuens dalam penelitian ini.
 Penjajaran sekuens jamak dilakukan menggunakan perangkat lunak geneious prime ( https://www.geneious.com ), seperti yang digambarkan dalam Gambar 1. Sekuens terkonservasi dari keluarga DUF152 diturunkan dari NCBI Conserved Protein Domain Family (pfam02578), dan Row Display digunakan ‘maks. 100 baris’ [ [ 46 ] ]. Identitas antara sekuens ini dapat divisualisasikan langsung dalam Tabel 2. LMCO bakteri ini tidak memiliki sekuens HXH terkonservasi yang biasanya digunakan oleh lakase untuk mengikat ion tembaga dan memfasilitasi transfer elektron. Perlu dicatat bahwa Lac13 memiliki sekuens N-terminal yang diperluas. Selain itu, prediksi domain struktural transmembran menggunakan alat TMHMM mengungkapkan tidak adanya domain tersebut dalam sekuens [ [ 47 , 48 ] ].
Penjajaran sekuens jamak dilakukan menggunakan perangkat lunak geneious prime ( https://www.geneious.com ), seperti yang digambarkan dalam Gambar 1. Sekuens terkonservasi dari keluarga DUF152 diturunkan dari NCBI Conserved Protein Domain Family (pfam02578), dan Row Display digunakan ‘maks. 100 baris’ [ [ 46 ] ]. Identitas antara sekuens ini dapat divisualisasikan langsung dalam Tabel 2. LMCO bakteri ini tidak memiliki sekuens HXH terkonservasi yang biasanya digunakan oleh lakase untuk mengikat ion tembaga dan memfasilitasi transfer elektron. Perlu dicatat bahwa Lac13 memiliki sekuens N-terminal yang diperluas. Selain itu, prediksi domain struktural transmembran menggunakan alat TMHMM mengungkapkan tidak adanya domain tersebut dalam sekuens [ [ 47 , 48 ] ].
Tabel 2. Identitas antara urutan Lac1 hingga Lac13.
 Urutan GGVS…H (residu histidin ini diberi label biru), yang sebagian ada, seperti yang ditunjukkan pada Gambar 1 , mungkin terlibat dalam pengikatan ion tembaga T2 atau T3. Urutan QXH…ADCXPVV…HAGWRG dapat ditegaskan sebagai situs pengikatan T1 Cu, di mana pola H…C…H cocok dengan situs pengikatan T1 Cu dari lakase khas. Urutan EV dalam beberapa protein dapat membentuk situs pengikatan ion tembaga T2 atau T3 dengan residu His, yang tidak menunjukkan identitas apa pun dengan urutan dari spesies lain dalam famili ini. Motif FY(YF/FF)SXRR(L) dari semua protein yang termasuk dalam kelompok ini terlibat dalam pembentukan kantong substrat dan, seperti yang ditunjukkan dalam hasil docking pada Gambar 2 , residu Arg dari beberapa protein mungkin terlibat dalam pengikatan substrat. Seperti yang ditunjukkan pada Gambar 1 , urutan protein yang dilestarikan yang digunakan dalam penelitian ini memiliki tingkat tumpang tindih yang tinggi dengan urutan DSM5159 dan PsLac, serta urutan yang dilestarikan dari keluarga DUF152, dan terutama pada pasangan asam amino H…C…H dari situs pengikatan ion tembaga. Mengingat bahwa anotasi prokka 1.12.0 (x3) dari urutan ini menghasilkan lakase, ada kemungkinan juga bahwa fosforilase nukleosida purin memiliki kemampuan untuk mengkatalisis substrat lakase. Urutan ini dikenakan prediksi struktur dan docking.
Urutan GGVS…H (residu histidin ini diberi label biru), yang sebagian ada, seperti yang ditunjukkan pada Gambar 1 , mungkin terlibat dalam pengikatan ion tembaga T2 atau T3. Urutan QXH…ADCXPVV…HAGWRG dapat ditegaskan sebagai situs pengikatan T1 Cu, di mana pola H…C…H cocok dengan situs pengikatan T1 Cu dari lakase khas. Urutan EV dalam beberapa protein dapat membentuk situs pengikatan ion tembaga T2 atau T3 dengan residu His, yang tidak menunjukkan identitas apa pun dengan urutan dari spesies lain dalam famili ini. Motif FY(YF/FF)SXRR(L) dari semua protein yang termasuk dalam kelompok ini terlibat dalam pembentukan kantong substrat dan, seperti yang ditunjukkan dalam hasil docking pada Gambar 2 , residu Arg dari beberapa protein mungkin terlibat dalam pengikatan substrat. Seperti yang ditunjukkan pada Gambar 1 , urutan protein yang dilestarikan yang digunakan dalam penelitian ini memiliki tingkat tumpang tindih yang tinggi dengan urutan DSM5159 dan PsLac, serta urutan yang dilestarikan dari keluarga DUF152, dan terutama pada pasangan asam amino H…C…H dari situs pengikatan ion tembaga. Mengingat bahwa anotasi prokka 1.12.0 (x3) dari urutan ini menghasilkan lakase, ada kemungkinan juga bahwa fosforilase nukleosida purin memiliki kemampuan untuk mengkatalisis substrat lakase. Urutan ini dikenakan prediksi struktur dan docking.
 Banyak sekuens yang dianotasi sebagai lakase juga dianotasi sebagai YfiH dan DUF152. Semua enzim dalam subfamili ini memiliki pasangan Cys-His. [ [ 45 ] ] Ini mirip dengan situs pengikatan ion tembaga dari lakase. DUF152 diklasifikasikan sebagai subfamili lakase dalam beberapa kasus. [ [ 4 ] ] Berdasarkan penyelarasan sekuens, sekuens LMCO yang dilestarikan yang digunakan dalam penelitian ini pada dasarnya sama dengan sekuens LMCO yang dilestarikan dari keluarga DUF152. Oleh karena itu, LMCO dalam penelitian ini seharusnya termasuk dalam keluarga DUF152.
Banyak sekuens yang dianotasi sebagai lakase juga dianotasi sebagai YfiH dan DUF152. Semua enzim dalam subfamili ini memiliki pasangan Cys-His. [ [ 45 ] ] Ini mirip dengan situs pengikatan ion tembaga dari lakase. DUF152 diklasifikasikan sebagai subfamili lakase dalam beberapa kasus. [ [ 4 ] ] Berdasarkan penyelarasan sekuens, sekuens LMCO yang dilestarikan yang digunakan dalam penelitian ini pada dasarnya sama dengan sekuens LMCO yang dilestarikan dari keluarga DUF152. Oleh karena itu, LMCO dalam penelitian ini seharusnya termasuk dalam keluarga DUF152.
Prediksi struktur tiga dimensi
Struktur tiga dimensi dari kelompok LMCO bakteri ini diprediksi dari urutan asam amino menggunakan alphafold 2. Seperti yang ditunjukkan pada Gambar 3 , badan utama dari struktur 3D kelompok ini menunjukkan tingkat tumpang tindih yang tinggi. Hasil ini konsisten dengan fakta bahwa, meskipun identitas urutan asam amino rendah antara LMCO ini, mereka memiliki struktur molekul yang serupa, dan geometri keseluruhan dari situs aktif mereka sangat kekal. [ [ 5 ] ] Ini memberikan dasar yang cukup untuk menyaring LMCO berdasarkan struktur tiga dimensi.
 Gambar 3
Gambar 3
Buka di penampil gambar
Kekuatan Gambar
Struktur 3D LMCO (Lac1 hingga Lac13) diprediksi oleh alphafold 2. Meskipun semua LMCO ini menunjukkan identitas urutan asam amino yang rendah, keseluruhan strukturnya masih menunjukkan tingkat kesamaan yang tinggi. Lembaran β sentral dari semua protein tumpang tindih secara signifikan, dan heliks di sekitarnya serta tren struktural keseluruhan sebagian besar tetap konsisten. Semua struktur diprediksi oleh alphafold 2 dengan ‘-model_preset=mono_casp14’.
Karena keuntungan lakase jamur untuk berbagai aplikasi, lakase bakteri dan MCO, terutama yang memiliki massa molekul rendah, masih relatif kurang dieksplorasi. Basis data PDB RCSB (https://www.rcsb.org) ditelusuri secara terpisah untuk enzim yang termasuk dalam kelas MCO, seperti yang ditunjukkan pada Tabel 3. Jumlah hasil menurun drastis ketika istilah pencarian tambahan untuk bakteri dan massa molekul di bawah 30 kDa diterapkan. Namun, massa molekul MCO yang digunakan dalam penelitian ini berada dalam kisaran 24 hingga 28,5 kDa. Baik pencarian urutan serupa oleh Uniprot blast , seperti yang ditunjukkan pada Tabel 1 , dan pencarian struktur serupa oleh PDB gagal menemukan MCO yang teridentifikasi. Ini menyoroti kesulitan dalam memperoleh informasi yang cukup dari hasil eksperimen dan basis data yang ada. Prediksi struktural memberikan lebih banyak informasi struktural. Ini memberikan kemungkinan untuk docking berikutnya.
Tabel 3. Hasil pencarian dalam basis data RCSB PDB.
 Docking ABTS
Docking ABTS
Penyaringan primer oleh autodock-vina dan rosetta-highresdocker mengungkapkan hasil berikut:
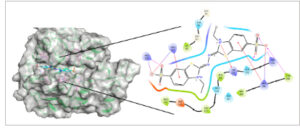
Semua urutan yang ditunjukkan pada Gambar 4 menunjukkan kantong substrat yang berbeda.
Kantong substrat dari semua enzim tumpang tindih dengan posisi ion tembaga T1. Hal ini juga membuktikan kebenaran simulasi ion tembaga T1 oleh metal3d karena reaksi oksidasi mirip lakase mengharuskan substrat kehilangan elektron di dekat ion tembaga T1. Oleh karena itu, ion tembaga T1 harus berada di wilayah yang sama dengan situs pengikatan substrat.
Asam amino terlibat dalam pembangunan langsung kantong substrat pada kira-kira setiap 20 asam amino yang dimulai pada asam amino N-terminal ~30.
Dibandingkan dengan lakase biasa, jenis LMCO ini, dengan panjang penuh sekitar 270 asam amino, relatif pendek, dan integritasnya sangat penting untuk konstruksi kantong substrat serta untuk aktivitas enzim.
Seperti yang ditunjukkan pada Gambar 1 dan 2 , beberapa protein menunjukkan bahwa ABTS dapat membentuk ikatan hidrogen dengan R pertama dalam urutan asam amino FY(YF/FF)SXRR(L) di ujung C-terminal, yang memberikan ide untuk desain LMCO baru untuk meningkatkan afinitas terhadap substrat seperti ABTS.
 Gambar 4
Gambar 4
Buka di penampil gambar
Kekuatan Gambar
Hasil docking Lac1 ke Lac13 dan Lac5-6 dengan ABTS. Ikatan hidrogen yang diprediksi ditunjukkan dengan garis putus-putus berwarna merah. Residu asam amino yang dapat membentuk ikatan hidrogen dengan ABTS ditunjukkan dengan warna ungu. Docking dilakukan dengan menggunakan rosetta-highresdocker dengan ‘-nstruct 50 000’ dan pymol digunakan untuk menggambar.
Docking ion tembaga
Prediksi pengikatan tembaga yang diperoleh dari metal3d menunjukkan hasil berikut:
Ikatan tembaga keseluruhan sangat bervariasi dari urutan ke urutan.
Sebagian besar sekuens memiliki satu ion tembaga dengan metrik keyakinan lebih besar dari 0,85, yang posisinya tumpang tindih dengan kantong pengikat substrat, sehingga sesuai dengan ion tembaga T1.
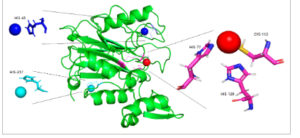
Situs pengikatan ion tembaga T1 berada di dekat N-terminal 70, Cys di dekat N-terminal 110, dan di dekat N-terminal 125. Seperti yang ditunjukkan pada Gambar 5 , Lac3 berfungsi sebagai contoh representatif.
 Gambar 5
Gambar 5
Buka di penampil gambar
Kekuatan Gambar
Hasil docking Lac3–Cu; warna merah menunjukkan ion tembaga T1. Docking dilakukan menggunakan metal3d dengan parameter default dan pymol digunakan untuk menggambar.
Pemilihan urutan LMCO
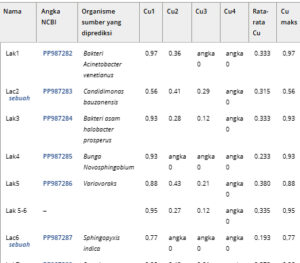
Hasil simulasi disajikan dalam Tabel 4. Karena ada empat ligan ion tembaga yang hadir dalam molekul lakase yang khas, penilaian terhadap ligan ion tembaga dibagi menjadi dua bagian: metrik keyakinan pengikatan ion tembaga pada posisi optimal dan metrik keyakinan pengikatan rata-rata dari empat ion tembaga. Urutan yang menunjukkan perbedaan signifikan antara hasil metal3d dan autodock-vina dalam hal docking ion tembaga dan celah besar antara hasil rosetta dan autodock-vina dalam hal docking substrat dihilangkan. Untuk docking ion tembaga, hasil metal3d digunakan. Untuk docking substrat, hasil rosetta digunakan. Sepuluh urutan dipilih secara acak yang memenuhi kondisi berikut.
Metrik keyakinan pengikatan ion tembaga pada posisi terbaik lebih besar dari 0,85.
Metrik keyakinan pengikatan ion tembaga rata-rata lebih besar dari 0,2.
Interface_delta_X kurang dari −16.
Tabel 4. Skor simulasi untuk urutan yang dipilih.
 Pada saat yang sama, dua sekuens dipilih secara acak dari sekuens yang tidak memenuhi kondisi di atas, dan sekuens Lac6, yang memiliki delesi yang jelas pada N-terminus, digunakan sebagai kontrol negatif. Semua sekuens diunggah ke NCBI, seperti yang ditunjukkan pada Tabel 4. Ujung N-terminal Lac5 dipilih untuk disambung dengan Lac6 setelah simulasi komputer, dan konstruksi yang dihasilkan diberi nama Lac5-6. Fragmen DNA mereka diperoleh menggunakan PCR fusi.
Pada saat yang sama, dua sekuens dipilih secara acak dari sekuens yang tidak memenuhi kondisi di atas, dan sekuens Lac6, yang memiliki delesi yang jelas pada N-terminus, digunakan sebagai kontrol negatif. Semua sekuens diunggah ke NCBI, seperti yang ditunjukkan pada Tabel 4. Ujung N-terminal Lac5 dipilih untuk disambung dengan Lac6 setelah simulasi komputer, dan konstruksi yang dihasilkan diberi nama Lac5-6. Fragmen DNA mereka diperoleh menggunakan PCR fusi.
Pengukuran aktivitas enzim
Seperti yang ditunjukkan pada Tabel 5 , semua sekuens dengan skor tinggi menunjukkan berbagai tingkat aktivitas enzimatik, sedangkan sekuens dengan skor rendah tidak menunjukkan aktivitas apa pun. Ini menunjukkan korelasi yang kuat antara penilaian komputasional dan aktivitas enzim aktual. Lac5-6 menunjukkan aktivitas yang lebih unggul dibandingkan dengan kedua protein asli, sejalan dengan data yang diprediksi. Ini menunjukkan efektivitas prediksi komputasional dalam mengidentifikasi sekuens dengan kinerja enzimatik yang ditingkatkan. Kolom kromatografi afinitas nikel yang digunakan untuk pemurnian LMCO ini menunjukkan penurunan warna yang nyata dan, setelah penggunaan berikutnya, efikasi pemurnian menurun secara signifikan. Sangat mungkin bahwa LMCO ini memperoleh beberapa ion nikel dari kolom afinitas selama proses pemurnian, sehingga mengakibatkan hilangnya aktivitas enzim. Enzim tersebut terbukti memiliki sifat kuat untuk mengikat ion logam divalen lainnya. Lac1 dan Lac3 menunjukkan aktivitas enzimatik yang berkurang dalam sistem reaksi yang kekurangan ion tembaga tambahan. Ini mungkin disebabkan oleh hilangnya ion tembaga selama proses pemurnian.
Tabel 5. Pengukuran aktivitas enzim. Uji aktivitas dilakukan menggunakan enzim murni pada konsentrasi akhir 100 m m buffer asetat dengan 2,5 m m tembaga sulfat, menggunakan ABTS sebagai substrat.
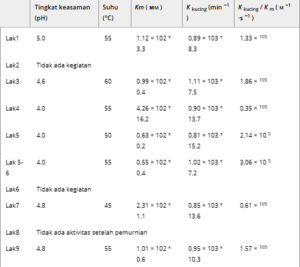 Sembilan protein yang diperoleh menunjukkan aktivitas yang lebih unggul dalam kondisi asam. Kelompok protein ini menunjukkan sedikit perbedaan antara hasil ABTS dan docking protein, seperti yang ditunjukkan pada Tabel 4. Pengikatan ion tembaga T1 ke protein muncul sebagai faktor penting yang memengaruhi aktivitas enzim. Parameter ini harus diprioritaskan sebagai kriteria utama untuk pemilihan LMCO.
Sembilan protein yang diperoleh menunjukkan aktivitas yang lebih unggul dalam kondisi asam. Kelompok protein ini menunjukkan sedikit perbedaan antara hasil ABTS dan docking protein, seperti yang ditunjukkan pada Tabel 4. Pengikatan ion tembaga T1 ke protein muncul sebagai faktor penting yang memengaruhi aktivitas enzim. Parameter ini harus diprioritaskan sebagai kriteria utama untuk pemilihan LMCO.
Selain itu, banyak faktor lain yang memengaruhi aktivitas lakase, seperti pengikatan ion tembaga, transpor elektron, dan pengikatan substrat, di antara banyak faktor lainnya. Aktivitas sembilan MCO diukur: lima MCO menunjukkan korelasi yang nyata antara hasil docking substrat dan K m , dengan koefisien korelasi sebesar 0,69, seperti yang ditunjukkan pada Gambar 6. Ketika kesembilan MCO dipertimbangkan, koefisien korelasinya masih 0,45. Ini juga dapat digunakan untuk menyaring lakase untuk aktivitas potensial terhadap substrat tertentu.
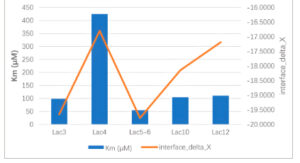 Gbr. 6
Gbr. 6
Buka di penampil gambar
Kekuatan Gambar
Hasil docking substrat dengan K m ; hasil docking berwarna oranye dan K m berwarna biru. Data untuk interface_delta_X berasal dari docking oleh rosetta-highresdocker dengan ‘-nstruct 50 000’. Data untuk K m adalah rata-rata dari tiga pengukuran.
Penutup
Dalam penelitian ini, kami mengambil 34 kemungkinan LMCO bakteri dari sekuens metagenom air limbah industri. Satu set 10 LMCO baru diidentifikasi menggunakan teknologi informasi untuk memprediksi struktur tiga dimensi, pengikatan tembaga dan pengikatan substrat, serta untuk memperbaiki satu protein yang kekurangan sekuens. Kelompok LMCO ini tidak menunjukkan kesamaan sekuens dengan lakase atau MCO yang ditinjau sebelumnya, dan mereka memiliki aktivitas optimal dalam lingkungan asam. Hasil pengukuran aktivitas enzim konsisten dengan yang diperoleh dari simulasi komputer. Akhirnya, penelitian ini menunjukkan nilai menggabungkan teknologi sekuensing metagenomik dengan pemodelan struktur protein dalam pencarian enzim baru, serta dalam penyaringan dan desain enzim baru. Kombinasi beberapa jenis perangkat lunak simulasi struktur protein dapat mengotomatiskan proses dari sekuens yang berasal dari metagenom hingga prediksi aktivitas dan spesifisitas enzim.
