Abstrak
Karsinoma ovarium serosa tingkat tinggi (HGSOC) dikaitkan dengan hasil pasien terburuk. Memahami lingkungan tumor dalam hal mengukur retrovirus endogen ( ERV ) dan ekspresi LINE-1 dan korelasinya dengan gen inflamasi, penghambat titik pemeriksaan, dan kelangsungan hidup pasien diperlukan. Analisis 102 HGSOC naif pengobatan dan jaringan kontrol untuk ERV , LINE-1 , inflamasi, dan titik pemeriksaan imun mengidentifikasi lima klaster dengan kelangsungan hidup bebas kekambuhan pasien yang beragam. Satu klaster yang disebut Triple-I dengan kelangsungan hidup pasien terbaik menunjukkan jumlah limfosit yang menyusup ke tumor tertinggi bersama dengan 22 gen yang diekspresikan berlebihan, termasuk CXCL9 dan AIM2 . Namun, Triple-I dikaitkan dengan ekspresi ERV/LINE-1 terendah . Klaster tumor dengan kelangsungan hidup pasien terbaik kedua memiliki ekspresi ERV/LINE-1 dan inflamasi yang tinggi. Imunohistokimia multipleks mengungkap protein CD28 hanya pada sel imun, tanpa ko-ekspresi reseptor penghambat CTLA4. Kelompok tumor terbesar dengan ekspresi ERV/LINE-1 yang tinggi tetapi peradangan rendah menunjukkan ekspresi gen yang rendah secara signifikan dari sensor dsRNA MDA5 dan RIG-I yang mendukung blok abnormal dalam pensinyalan IFN. Studi kami merupakan ‘gambaran molekuler dan imunologis’ intrinsik dari lingkungan tumor HGSOC yang penting untuk memahami retroelemen dan peradangan untuk relevansi klinis.
Singkatan
TUJUAN 2
tidak ada pada melanoma
Bahasa Indonesia: CXCL9
Ligand kemokin motif CXC
DEG
gen yang diekspresikan secara berbeda
lingkungan
amplop
Bahasa Indonesia: ERV
retrovirus endogen
HGSOC
karsinoma ovarium serosa tingkat tinggi
INF
peradangan
IRGS
peradangan dan tanda tangan gen pengatur
L1-5′UTR
LINE-1 5′ daerah yang belum diterjemahkan
L1-ORF2
LINE-1 bingkai baca terbuka 2
GARIS-1
unsur nuklir berselaput panjang-1
Bahasa Indonesia: RFS
kelangsungan hidup bebas kekambuhan
STIL
tumor stroma yang menyusup ke limfosit
WAKTU
lingkungan mikro imun tumor
1 Pendahuluan
Tingkat peradangan tumor yang rendah atau tinggi menentukan kelangsungan hidup pasien [ [ 1 ] ]. Peradangan berasal dari aktivasi dan perekrutan respons imun bawaan dan adaptif. Presentasi antigen, bersama dengan reseptor titik pemeriksaan imun dan interaksi ligan sel imun, merupakan bagian normal dari respons imun atau dapat menyimpang karena respons adaptif dari lingkungan mikro imun tumor (TIME). Setelah induksi respons imun bawaan dan pensinyalan interferon (IFN) tipe I, kemungkinan interaksi silang dengan respons imun adaptif dapat terjadi [ [ 2 ] ]. Misalnya, setelah aktivasi reseptor tol bawaan (TLR), reseptor mirip NOD (NLR), reseptor mirip RIG-I (RLR) dan gen yang terstimulasi IFN seperti IFI16, reseptor mirip AIM2, bersama dengan sitokin (misalnya, IL18) ekspresi IFNG menjadi terinduksi. IFNG disekresikan oleh sel imun seperti sel pembunuh alami (NK), limfoid, sel T helper, dan sel T sitotoksik [ [ 3 ] ]. Selain pembunuhan langsung sel T sitotoksik terhadap sel yang terinfeksi virus atau sel tumor, sinyal IFN pada akhirnya menghasilkan apoptosis atau piroptosis melalui inflamasom [ [ 4 ] ].
Beberapa asal peradangan ada. Misalnya, ‘peradangan steril’ terjadi tanpa adanya mikroorganisme dan disebabkan oleh sinyal endogen seperti protein syok panas, RNA atau DNA yang dikenali oleh TLR dan sensor lainnya [ [ 5 ] ]. Salah satu sumber penting RNA yang menginduksi peradangan tumor adalah retrovirus endogen ( ERV ) [ [ 6 , 7 ] ]. ERV adalah bagian dari keluarga retroelemen pengulangan terminal panjang (LTR) yang terintegrasi di seluruh genom manusia dari infeksi masa lalu [ [ 8 ] ]. ERV adalah elemen berulang dan mewakili 5–8% genom, meskipun jumlah totalnya bervariasi karena bentuk mosaik [ [ 9 ] ]. ERV diklasifikasikan menjadi kelas I (gamma/epsilon), kelas II (alfa/beta) retrovirus, dan kelas III atau spumavirus. Beberapa kelompok famili ERV ada, di mana ERV-K adalah yang paling banyak terwakili [ [ 10 ] ]. Meskipun sebagian besar elemen ERV rusak karena mutasi dan deleksi dari waktu ke waktu, banyak elemen ERV yang mempertahankan fungsionalitas protein, khususnya protein envelope (env) [ [ 11 ] ] (Gbr. S1A ). Secara umum, ERV dibungkam dalam jaringan somatik karena regulasi epigenetik. Namun, ekspresi ERV menjadi tidak ditekan selama perkembangan [ [ 12 ] ] dan dalam berbagai tumor [ [ 13 , 14 ] ] serta dalam penentuan stadium tumor [ [ 15 ] ]. Yang penting, peran protein ERV sebagai antigen tumor, dengan fungsi pendorong tumor, dapat mewakili target terapi baru, yang sedang diselidiki [ [ 16 , 17 ] ]. Aktivasi elemen ERV tumor yang terjadi secara intrinsik atau setelah pengobatan penghambat epigenetik, yang didefinisikan sebagai mimikri virus, menghasilkan kompleks RNA untai ganda (ds) ERV dan menginduksi sinyalisasi IFN tipe I melalui imunitas bawaan [ [ 6 , 7 , 18 ] ].
Elemen nuklir berseling panjang ( LINE ) -1 (L1) adalah retrotransposon non-LTR dan telah mengakumulasi mutasi dan penghapusan selama evolusi, yang mengakibatkan khususnya pemotongan 5′UTR (untuk tinjauan [ [ 19 ] ]). L1 mewakili lebih dari setengah juta elemen berulang yang cacat di seluruh genom manusia. Elemen berulang L1 yang cacat ini dapat ditranskripsi dan berpartisipasi dalam kontrol regulasi perilaku seluler yang dimediasi RNA. Sebaliknya, hanya ada ~ 100 elemen L1 panjang penuh yang aktif , yang mengkode protein yang terlibat dalam retrotransposisi aktif [ [ 20 ] ]. L1 panjang penuh terdiri dari daerah tidak diterjemahkan 5′ (UTR), yang menampung promotor untuk transkripsi dan sinyal inisiasi transkripsi antisense untuk kerangka baca terbuka (ORF)0, serta dua kerangka baca terbuka ( ORF ) -1 dan ORF2 , dan 3′UTR (Gbr. S1A ). Yang penting, menggunakan pencitraan dan analisis biokimia menunjukkan bahwa protein reverse transcriptase L1 – ORF2 secara istimewa mengikat substrat RNA dan secara kuat menghasilkan hibrida RNA:DNA sitosolik [ [ 21 , 22 ] ]. Akumulasi hibrida RNA:DNA seluler menyebabkan aktivasi pensinyalan imun bawaan melalui cGAS/STING dan produksi IFN. Sebaliknya, subset L1 aktif yang diketahui , L1PA1 yang termasuk dalam subfamili L1Hs , dan L1PA2 adalah sumber dsRNA, yang menginduksi gen terstimulasi IFN (ISG) melalui pensinyalan JAK/STAT [ [ 23 ] ]. Meskipun ERV dan L1 memicu pensinyalan IFN, penelitian lebih lanjut diperlukan untuk mengukur tingkat ekspresi gen ERV dan L1 tumor intrinsik yang tinggi dan rendah dan bagaimana keduanya berkorelasi satu sama lain, serta dengan gen inflamasi, penghambat titik pemeriksaan, dan kelangsungan hidup pasien. Karena sifat ERV dan khususnya LTR yang sangat repetitif , masih menjadi tantangan untuk mengidentifikasi dan melokalisasi mereka secara akurat dalam genom menggunakan protokol RNA-seq standar, seperti keluarga ERV-K , yang mencakup lebih dari 550 provirus dan lebih dari 6400 LTR [ [ 24 ] ]. Misalnya, saat menggunakan RNA-seq, beberapa pembacaan pemetaan dapat mengakibatkan eliminasi ERV yang melimpah atau bahkan kehilangan ERV individual .elemen dan lokasinya saat menggunakan model empiris. Menerapkan pembacaan RNA-seq nukleotida pendek standar 76–150 untuk daerah berulang juga dapat menghasilkan banyak kecocokan dan dengan demikian melokalisasi ERV ke lokasi yang salah sehingga menghasilkan regulasi naik atau turun yang tidak akurat [ [ 25 , 26 ] ]. Meskipun demikian, sampel tumor pasien yang berbeda dianalisis untuk ERV menggunakan teknologi RNA-seq mengidentifikasi subfamili ERV umum , misalnya, LTR solo , MER , ERV-E , dan ERV-H dengan penekanan pada peradangan [ [ 27 – 29 ] ].
Karsinoma ovarium serosa tingkat tinggi (HGSOC) adalah kanker ovarium yang paling umum dan mematikan dengan kelangsungan hidup pasien yang buruk secara keseluruhan antara 2,8 dan 5,1 tahun tergantung pada limfosit yang menyusup ke tumor (TIL). HGSOC telah dihipotesiskan berasal dari tuba falopi atau epitel permukaan ovarium [ [ 30 – 32 ] ]. Pengobatan HGSOC terus menjadi sulit dan sejauh ini imunoterapi belum memenuhi harapan [ [ 33 ] ], meskipun adanya infiltrasi sel T CD8 + [ [ 34 ] ]. Penelitian lebih lanjut diperlukan untuk memahami molekul pengatur titik pemeriksaan dan biologinya dalam HGSOC TIME. Dalam penelitian ini, kami mengukur tingkat ekspresi intrinsik dari beberapa keluarga ERV kodogenik dan nonkodogenik , L1-5′UTR dan L1-ORF2 dalam 102 HGSOC pasien tanpa pengobatan sebelumnya. Metodologi kami menerapkan primer gen spesifik dan qPCR absolut, di mana tingkat ekspresi diukur sepenuhnya menggunakan gen kloning. Ekspresi ERV , L1-5′UTR dan L1-ORF2 berkorelasi secara signifikan satu sama lain dalam HGSOC dan dengan gen yang terkait dengan DNA hipometilasi pada kanker. Ekspresi gen ERV dan L1-ORF2 juga berkorelasi secara signifikan dengan gen inflamasi tumor, sel imun, gen pengatur titik pemeriksaan imun, dan kelangsungan hidup pasien. Kami mengidentifikasi gen penting untuk kelangsungan hidup serta menentukan regulasinya menggunakan lini sel HGSOC. Studi kami merupakan ‘gambaran molekuler’ intrinsik unik dari TIME HGSOC sebelum perawatan pasien untuk mendapatkan wawasan tentang retroelemen dan inflamasi untuk relevansi klinis. Kami menyajikan temuan molekuler baru mengenai biologi TIME yang penting untuk kelangsungan hidup pasien.
2 Bahan dan Metode
2.1 Data klinis dan patologis pasien, tumor, dan jaringan kontrol
Semua pengambilan sampel jaringan primer manusia dan penelitian blok tumor primer yang difiksasi formalin-tersimpan dalam parafin (FFPE) telah disetujui oleh Komite Etik Universitas Friedrich-Alexander Erlangen-Nuremberg, Jerman (#199_19Bc dan #252_20B) sesuai dengan Deklarasi Helsinki dari Asosiasi Medis Dunia. Eksperimen dilakukan dengan pemahaman dan persetujuan tertulis dari setiap subjek. Semua data klinis dan patologis pasien dikumpulkan dari Comprehensive Cancer Center (CCC), Erlangen (Tabel 1 ).
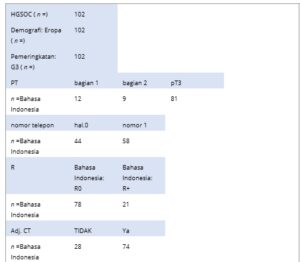
Tabel 1. Parameter klinis dan patologis dan status inflamasi dari kelompok tumor ERV-L1-ORF2 dari pasien dengan karsinoma ovarium serosa tingkat tinggi (HGSOC). Dari atas ke bawah: kohort HGSOC ( n = 102), dengan usia rata-rata 61 tahun. Semua pasien adalah orang Eropa di mana 96 pasien adalah Jerman dan enam pasien dengan Kebangsaan Polandia, Turki dan Serbia. Semua 102 tumor dinilai secara patologis sebagai G3 tingkat tinggi dengan stadium tumor pT: pT1-3; kelenjar getah bening negatif (pN0) atau pN1 positif; R = tumor residual, R0 = reseksi untuk tujuan kuratif, R+ = tumor residual ada; dan dari tiga pasien, tidak ada informasi yang tersedia. Mengenai kemoterapi adjuvan (adj.) (CT): 74 Pasien didaftarkan ke dalam program pengobatan terapi standar dengan: karboplatin dan paclitaxel ( n = 62); carboplatin, paclitaxel, dan bevacizumab ( n = 2); carboplatin, paclitaxel, dan gemcitabine ( n = 1); carboplatin ( n = 2); tujuh pasien tanpa informasi pengobatan yang tersedia; dan 28 pasien tidak menerima pengobatan kemoterapi; penampakan stroma tumor; sTIL = limfosit T yang menyusup ke stroma; ekspresi ERV-L1 (molekul·ng −1 RNA) untuk semua tumor, baik tinggi maupun rendah, ditentukan oleh qPCR absolut; lima kelompok tumor ditentukan oleh total molekul ERV-L1-ORF2 ·ng −1 RNA dibandingkan dengan 99 gen peradangan tumor yang didefinisikan sebagai tanda tangan gen pengatur peradangan (IRGS). Selain itu, kelangsungan hidup bebas kekambuhan hingga 66 bulan (5,5 tahun) tersedia untuk semua 102 pasien HGSOC. ERV, retrovirus endogen.
Jaringan yang diwarnai hematoxylin dan eosin (H&E) dari blok tumor FFPE primer yang diarsipkan ( n = 102) disaring oleh dua Ahli Patologi (ME, AH) untuk mengonfirmasi histologi HGSOC, gradasi (semua G3) dan kandungan tumor > 85%. Pasien dengan metastasis jauh tidak disertakan dalam penelitian ini. Usia rata-rata pasien adalah 61 tahun (Tabel 1 ). Semua jaringan HGSOC H&E dinilai untuk jenis stroma dan TIL stroma (sTIL) (Tabel 1 ). Tuba fallopi non-pasien sehat FFPE yang cocok ( n = 13) diperoleh dari wanita yang menjalani sterilisasi elektif. Jaringan ovarium yang sehat ( n = 16; usia rata-rata = 57 tahun) seperti yang sebagian dijelaskan sebelumnya [ [ 6 ] ], dibedah dari pasien dengan HGSOC (pasien yang cocok ( n = 2) dan tidak cocok dengan pasien ( n = 3)), wanita yang menjalani operasi untuk kista ovarium ( n = 7), dermoid ( n = 1) atau leiomyoma ( n = 1) serta dari pasien tanpa kondisi patologis ( n = 2).
2.2 Isolasi RNA untuk ekspresi gen dan hibridisasi RNA multipleks langsung (NanoString)
Semua protokol sesuai dengan Kohler et al. [ [ 35 ] ]. Untuk tumor dan jaringan sehat, potongan jaringan setebal 10 μm dari dua blok tumor berbeda per pasien digabungkan. Potongan jaringan diperlakukan dengan metilen biru, dan RNA diisolasi menggunakan kit Maxwell RSC RNA FFPE (Promega, #AS1440, Walldorf, Jerman). Setelah perlakuan DNase I, RNA dimurnikan dan diukur lebih lanjut. NanoString PlexSet dengan 260 gen dirancang dari penelitian kami sebelumnya dan dimodifikasi [ [ 35 , 36 ] ] (Tabel S1A ). 260 gen ini mencakup gen referensi yang diketahui serta gen yang penting untuk perkembangan siklus sel, remodeling matriks ekstraseluler (ECM), transisi epitel ke mesenkimal, fenotipe tumor, fenotipe sel imun, dan peradangan. 37 gen referensi umum yang dipilih dari literatur dianalisis dengan RNA tumor dan jaringan sehat menggunakan NanoString dan qPCR untuk menentukan gen referensi yang paling sesuai [ [ 35 ] ]. Menurut koefisien variasi (COV), dua gen, beta-aktin ( ACTB ; HGNC:132) dan Tyrosine 3-monooxygenase/tryptophan 5-monooxygenase activation protein zeta ( YWHAZ ; HGNC:12855) adalah yang paling konstan dalam ekspresinya untuk semua HGSOC dan tuba falopi normal (Gbr. S1B ). Setelah normalisasi RNA tumor dan jaringan sehat dengan ACTB dan YWHAZ , kami kemudian melakukan analisis spesifik gen dengan 99 gen yang dipilih dari panel gen NanoString, yang berkorelasi dengan respons imun (jalur IFN, sensor asam nukleat, fenotipe sel imun, dan sitokin serta pengatur imun lainnya). Panel gen terpilih yang berjumlah 99 yang disebutkan di seluruh naskah disebut sebagai Inflammation Regulatory Gene Signature (IRGS) (Tabel S1B ). Untuk validasi ekspresi gen NanoString dan qPCR (dijelaskan di bawah), kami memilih satu gen dari panel IRGS, ZBP1 , untuk konfirmasi (Tabel S1B,C , Gambar S1C,D ). Yang penting, hasil serupa ditemukan menggunakan kedua teknik ekspresi gen.
2.3 PCR kuantitatif dan real-time kuantitatif penuh dari ERV , L1-5′UTR dan L1-ORF2
Semua protokol untuk qPCR kuantitatif (q) dan absolut menggunakan cDNA dan primer ERV dan L1 yang kami publikasikan (Tabel S1C,D ) dan campuran utama SYBR select, dilakukan menggunakan sistem PCR StepOne plus menurut berbagai publikasi (Thermo Fisher, Waltham, MA, AS) [ [ 6 , 15 , 17 , 37 ] ]. QPCR dinormalisasi menggunakan dua gen referensi terbaik ACTB dan YWHAZ untuk HGSOC dan FT seperti dijelaskan di atas. Fragmen L1-ORF2 (660 bp) diamplifikasi menggunakan primer TF 5′ GAGGCCAGCATCATTCTGATACCAAAGCCG dan BR 5′ TCCTGAGACTTTGCTGAAGTTGCTTATCAGC dan kemudian dikloning ke dalam vektor pSC-A (Agilent, Waldbronn, Jerman). Menggunakan primer untuk qPCR L1-ORF2 [ [ 37 ] ] (Tabel S1C ), kami menghitung kemiringan y = −3,5537 + 38,997 dan R 2 sebesar 0,9908 menggunakan kurva standar dengan fragmen L1-ORF2 yang diklon . Selain itu, L1-5′UTR juga dianalisis secara kuantitatif sepenuhnya, di mana wilayah gen 5′UTR dari nukleotida 365 hingga 854 dipilih menurut elemen L1 manusia LRE2 dari kromosom 1q (U09116.1). Wilayah L1 -5′UTR ini disintesis (BioCat, Heidelberg, Jerman) dan diklon ke dalam pBlueScript II KS(+) (Agilent Tech., AS). Menggunakan primer untuk L1-5′UTR [ [ 38 ] ] (Tabel S1C ), kami menghitung kemiringan y = −3,129 + 39,942 dan R 2 sebesar 0,9973 menggunakan kurva standar dengan fragmen L1-5′UTR yang diklon . Semua nilai ekspresi qPCR ERV , L1-5′UTR dan L1-ORF2 ditransformasikan menjadi molekul·ng −1 total RNA menggunakan kurva standar dengan nilai ambang siklus (cT) terhadap log jumlah standar yang menerapkan nomor salinan yang diketahui dari setiap gen yang diklon sebagai standar eksternal [ [ 15 ] ]. Total ERV menunjukkan ekspresi gabungan dari semua ERV yang dipelajari dalam penyelidikan ini (Tabel S1C,D ). Kami juga menyertakan gen yang diketahui untuk de-represi genom sebagai kontrol PCR. Kami memilih faktor pengikat antigen kanker testis CCCCTC seperti ( CTCFL , juga disebut BORIS ), yang mengalami hipometilasi pada beberapa kanker [ [ 39 ]], tetapi terutama di HGSOC dibandingkan dengan jaringan kontrol [ [ 40 ] ]. Semua primer qPCR tambahan dijelaskan dalam Tabel S1C .
2.4 Imunohistokimia (IHC) sel imun
Sembilan antibodi yang sebelumnya dijelaskan, CD4, CD8, FOXP3, LAG3, CD80, PD1, PD-L1, CTLA4, dan MHC-I, khusus untuk penanda permukaan sel dihibridisasi ke jaringan mikroarray (TMA) yang baru dibangun yang mewakili semua 102 HGSOC di Institut Patologi untuk mengidentifikasi sel-sel imun yang berbeda dan penghambat titik pemeriksaan imun atau molekul protein reseptor menurut Pfannstiel et al. [ [ 41 ] ]. Untuk CD28 (klon antibodi RM404, Novus Biologicals, Centennial, CO, AS) pengenceran 1: 1000 dihibridisasi ke TMA di atas dan jaringan kelenjar getah bening yang cocok dengan pasien ( n = 14) bersama dengan antibodi CTLA4. Untuk Multiplex IHC, baik antibodi CTLA-4 (klon BSB-88, dari Bio SB, Medac GmbH, Wedel, Jerman) dan CD28 dihibridisasi ke potongan jaringan HGSOC ( n = 9). Singkatnya, setelah rehidrasi jaringan, sampel diinkubasi dalam buffer TRIS pH 6 selama 15 menit; kemudian, antibodi CTLA4 (1 : 200) diinkubasi semalam pada suhu 4 °C. Untuk mendeteksi CTLA4, polimer alkaline phosphatase (AP) dengan permanent red (ZytoChem Plus (AP) Polymer Kit’, POLAP-100, dari Zytomed, Berlin, Jerman) digunakan. Antibodi CD28 (1 : 500) dihibridisasi selama 2 jam pada suhu RT dan kemudian dideteksi menggunakan Polymer-AP dan Permanent Green (Zytomed). Terakhir, semua 102 jaringan yang diwarnai HGSOC H&E dianalisis (ME; LG) untuk mengukur persentase sTIL. STIL dinilai dalam stroma tumor menurut metodologi yang divalidasi dari International Working Group on TILs seperti yang dijelaskan dalam Pfannstiel et al. [ [ 41 ] ].
2.5 Studi kultur sel
Dua lini sel HGSOC, TyKnu (RRID:CVCL_1776) dan Kuramochi (RRID:CVCL_1345), diperoleh setelah karakterisasi ekspresi genomik dan RNA [ [ 42 ] ] serta autentikasi dari Profesor S. Baylin, Universitas Johns Hopkins, AS. Lini sel TyKnu dan Kuramochi diautentikasi menggunakan 20 penanda STR (short tandem repeat) dan menunjukkan 100% identitas dengan penanda sel referensi menurut Papp et al. [ [ 42 ] ] (lihat Tabel S1 di dalam). Semua percobaan kultur sel dilakukan dalam waktu 3 tahun setelah autentikasi. Kedua lini sel dipelihara dalam media RPMI 1640 yang dilengkapi dengan 10% FCS, 1 × NEAA (Thermo Fisher), l -glutamin (Thermo Fisher) pada suhu 37 °C, 5% CO 2 , dan dianalisis secara rutin untuk mikoplasma. Semua percobaan dengan lini sel di atas dilakukan dengan sel bebas mikoplasma. Lini sel diobati dengan IFNG (R&D Systems, Minneapolis, MN, AS) (10 dan 50 ng·mL −1 ) selama 21 jam, dan kemudian, RNA diekstraksi dengan TriFast (VWR, Darmstadt, Jerman) sesuai dengan rekomendasi pabrik pembuatnya. qPCR waktu nyata untuk CXCL9 dan AIM2 dilakukan seperti yang dijelaskan di atas (lihat Tabel S1C untuk primer).
2.6 Analisis bioinformatika
Perangkat lunak komputer sas jmp , SAS Inst., Heidelberg, Jerman 13.2 digunakan untuk membuat peta panas ekspresi gen, analisis kelangsungan hidup pasien Kaplan–Meier, dan analisis regresi COX [ [ 41 ] ]. Ekspresi protein spesifik yang direpresentasikan oleh setiap hibridisasi IHC dikuantifikasi menggunakan perangkat lunak qupath menurut Pfannstiel et al. [ [ 41 ] ] untuk menganalisis pusat tumor, bagian depan invasi, dan kemudian menghitung jumlah rata-rata. Perangkat lunak komputer qupath V0.5.0 digunakan bersama dengan alat pelatihan kecerdasan buatan (AI) untuk kuantifikasi sel CD28 + (hijau) dan CTLA4 + (merah) positif ganda dan sinyal tunggal untuk sel CD28 + (hijau) menggunakan Multiplex IHC. Lokalisasi semua pasangan primer ERV dan L1 pada kromosom manusia dilakukan dengan Blast® ( https://blast.ncbi.nlm.nih.gov/Blast.cgi ). Untuk pasangan primer ERV , ditetapkan identitas ≥ 94%, 0 celah, dan skor 38,3–44,6 bit, yang memungkinkan 1 atau 2 ketidakcocokan dalam urutan primer, serta untuk identitas 100% (Tabel S1E ). Kami juga melakukan analisis kromosom in silico dari pasangan primer L1-5′UTR dan L1-ORF2 dengan identitas 100% (TF dan BR) dan panjang amplikon yang identik menggunakan peramban penyelarasan kromosom NCBI: https://www.ncbi.nlm.nih.gov/gdv/browser/genome/?id=GCF_000001405.40&chr=1 . Lokalisasi nukleotida yang tepat dari setiap pasangan primer L1-5′UTR dan L1-ORF2 menggunakan rakitan primer referensi GRCh38.p14 dengan rilis anotasi RS_2024_08 (Tabel S1E ) diidentifikasi (Tabel S1F,G ).
Bahasa Indonesia: Untuk mengidentifikasi gen yang diekspresikan secara berbeda secara signifikan (DEG) antara klaster inflamasi HGSOC ERV-L1-ORF2 menggunakan nilai ekspresi total NanoString atau IRGS, uji statistik tertentu, seperti deseq 2 (jalur analisis default yang memerlukan data hitungan mentah NanoString sebagai input), uji- t Welch untuk memperoleh per gen, nilai- P yang tidak disesuaikan untuk perubahan log2-lipat dan prosedur Benjamini–Hochberg untuk menyesuaikan nilai- P untuk beberapa pengujian digunakan. Uji statistik ini menghasilkan tingkat signifikansi yang ditetapkan sebagai P < 0,01 dengan perbedaan minimal 2 dalam log2. CIBERSORT [ [ 43 ] ] mengidentifikasi sel imun yang berasosiasi dengan lima klaster tumor inflamasi /ERV-L1-ORF2 . Untuk memperoleh kelimpahan total berbagai jenis sel imun, ditetapkan sebagai nilai ekspresi absolut berdasarkan 225 ekspresi gen NanoString (223 gen plus ACTB dan YWHAZ ); nilai dirata-ratakan dari semua klaster tumor, termasuk tuba fallopi. Normalisasi terhadap kelompok tumor radang individual menghasilkan populasi sel imun terpisah dalam persen. Kelimpahan jenis sel imun dalam lima kelompok tumor radang dibandingkan dengan tuba fallopi diperoleh dengan mengukur perubahan log2-fold.
Pendekatan pemangkasan digunakan untuk menentukan set gen minimal yang mengklasifikasikan lima klaster tumor inflamasi ERV-L1-ORF2 yang berbeda , menggunakan set gen NanoString total atau panel IRGS NanoString beserta tingkat ekspresi gen ERV-L1-ORF2 individual . Prosedur pemangkasan dimulai dengan menyesuaikan model regresi logistik ke semua nilai ekspresi gen yang tersedia untuk memprediksi apakah setiap sampel merupakan bagian dari klaster yang dipilih (menggunakan validasi silang leave-one-out). Setelah penyesuaian model, pentingnya setiap gen ditentukan dan gen yang paling tidak penting dipangkas dari kumpulan data. Selanjutnya, model regresi logistik disesuaikan lagi menggunakan set nilai ekspresi gen yang lebih kecil, dan kemudian gen yang paling tidak penting dihilangkan. Set gen minimal diidentifikasi oleh model yang sesuai yang mencapai presisi tertinggi dalam mengklasifikasikan klaster yang benar (jika beberapa set gen mencapai presisi yang identik, kami memilih yang terkecil). Skor inflamasi dihitung untuk setiap sampel tumor dari klaster inflamasi rendah dan inflamasi tinggi sebagai jumlah tingkat ekspresi log2 dari 99 gen IRGS NanoString. Skor peradangan merangkum status peradangan HGSOC dalam satu metrik berkelanjutan. Skor ini secara langsung dibandingkan dengan nilai total ERV dan ekspresi L1-ORF2 untuk setiap kelompok peradangan.
3 Hasil
3.1 Ekspresi ERV , L1-5′ UTR , dan L1-ORF2 di jaringan HGSOC dan kontrol
Untuk mengukur ekspresi gen ERV , L1-5′UTR , dan L1-ORF2 dalam HGSOC dan jaringan sehat, kami menggunakan qPCR absolut yang dikembangkan sebelumnya oleh kami [ [ 6 , 14 , 15 ] ]. Metodologi ini memungkinkan perbandingan langsung molekul ERV dan L1 antara jaringan yang berbeda. Pasangan primer ERV yang kami rancang mengidentifikasi semua keluarga ERV utama dalam genom dengan jumlah kelompok yang rendah dan tinggi yang diklasifikasikan menurut keluarga LTR tertentu (Tabel S1C,D ). Misalnya, ERV-Fc , ERV-FRD , ERV-Fb , ERV-F(XA34) , ERV-H , ERV-W , ERV-K , ERV-R , ERV-E , ERV-Pb , ERV-T , ERV-Rb , MER34 , HRES1 , dan ERV9 , termasuk keluarga dengan beberapa kelompok pada kromosom yang berbeda, seperti ERV-K dengan 27 kelompok (Tabel S1C,D ). Menganalisis genom manusia untuk penyelarasan semua pasangan primer ERV dengan identitas 100% atau ≥ 94% menggunakan Blast® menghasilkan berbagai lokalisasi untuk setiap kromosom (Tabel S1E ). Misalnya, 11 (100% identitas) atau 75 (≥ 94% identitas) lokus kromosom yang berbeda ditemukan untuk pasangan primer ERV-Kenv , tetapi hanya satu lokasi untuk ERV-Rb (kromosom 3) menggunakan kedua penyelarasan. Penyelarasan kromosom pasangan primer L1-5′UTR dan L1-ORF2 dengan 100% identitas dan panjang amplikon yang identik menghasilkan 1281 dan 555 lokus, masing-masing (Tabel S1E–G ). Menariknya, hasil ini menunjukkan jumlah lokalisasi kromosom yang lebih tinggi untuk L1-5′UTR versus L1-ORF2 menggunakan urutan primer spesifik ini (Tabel S1F,G ). Meskipun beberapa publikasi telah menjelaskan pemotongan 5′ yang sering terjadi pada L1 (untuk tinjauan [ [ 19 ] ]), penting untuk dicatat bahwa jumlah lokalisasi kromosom L1-5′UTR yang lebih tinggi dibandingkan dengan L1-ORF2 diidentifikasi dengan pasangan primer spesifik ini menggunakan identitas 100% (Tabel S1C ). Dengan demikian, analisis genomik bioinformatika tambahan diperlukan terkait L1-5′UTR dan L1-ORF2 .
Selanjutnya kami mengukur keluarga ERV dan L1-5′UTR dan L1-ORF2 untuk tingkat ekspresi gen dalam HGSOC ( n = 102) dibandingkan dengan tuba fallopi yang sehat ( n = 13) dan jaringan ovarium ( n = 16) menggunakan urutan primer kami. Hasil penelitian menunjukkan bahwa elemen ERV , L1-5′UTR dan L1-ORF2 diekspresikan secara signifikan pada tumor dibandingkan dengan jaringan ovarium ( P <0,0001) (Gbr. 1A ). Anehnya, gen L1-ORF2 diekspresikan secara signifikan pada tuba fallopi dibandingkan dengan tumor (Gbr. 1A ). Yang penting, membandingkan tingkat ekspresi gen total HGSOC dari ketiga retroelemen menggunakan primer kami menunjukkan hierarki, di mana L1-5′UTR adalah yang tertinggi (rata-rata = 137 287,57 molekul·ng −1 RNA), kemudian L1-ORF2 (rata-rata = 61 280,27 molekul·ng −1 RNA) dan total ERV (rata-rata = 2422,68 molekul·ng −1 RNA) sebagai yang terendah. Tingkat ekspresi L1-5′UTR yang lebih tinggi 2 kali lipat dibandingkan L1 -ORF2 dapat berasal dari lebih banyak lokasi kromosom L1-5′UTR menggunakan primer kami (Tabel S1E ). Untuk mendapatkan wawasan, jika tingkat ekspresi gen HGSOC dari ERV , L1-5′UTR dan L1-ORF2 berkorelasi satu sama lain, kami melakukan analisis korelasi Spearman. Yang mengejutkan, hasil penelitian menunjukkan bahwa koefisien korelasi Spearman (IK 95%) sangat signifikan antara total ERV , L1-5′UTR dan L1-ORF2 dan berkisar antara r = 0,8188 dan 0,9853 (Gbr. S2 ). Untuk menguji lebih lanjut, apakah ekspresi total ERV , L1-5′UTR dan L1-ORF2 berkorelasi dengan de-represi genomik, kami menganalisis gen CTCFL , yang diketahui mewakili keadaan DNA hipometilasi pada kanker, termasuk HGSOC [ [ 40 ] ]. Hasil penelitian menunjukkan korelasi positif yang signifikan antara total ERV , L1-5′UTR dan ekspresi L1-ORF2 dengan CTCFL ( P < 0,0001) (Gbr. S2 ). Secara keseluruhan, kami menginterpretasikan temuan kami di atas bahwa ekspresi berlebihan ERV , L1-5′UTR dan L1-ORF2sangat terekspresi dan terkait erat dalam HGSOC, yang sebagian dapat dijelaskan karena hipometilasi DNA [ [ 6 , 15 , 40 , 44 ] ].
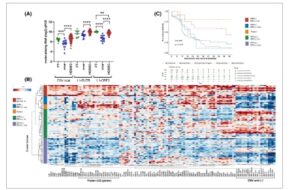
Gbr. 1
Buka di penampil gambar
Kekuatan Gambar
(A) Hasil ekspresi gen retroelemen karsinoma ovarium serosa tingkat tinggi (HGSOC) dari total retrovirus endogen ( ERV ), L1-5′UTR dan L1-ORF2 dibandingkan dengan jaringan kontrol tuba fallopi dan ovarium yang sehat. Hasil PCR real-time kuantifikasi absolut untuk setiap jaringan menunjukkan tingkat ekspresi gen (warna hijau = Tuba fallopi (FT) ( n = 13); biru = ovarium (ovar) ( n = 16) dan merah = HGSOC ( n = 102)). Total ERV menunjukkan ekspresi gabungan dari semua ERV yang dipelajari dalam investigasi ini (Tabel S1C,D ). Total kadar molekul·ng −1 RNA L1-5′UTR dan L1-ORF2 per jaringan juga ditunjukkan (Tabel S1C ). Grafik menunjukkan log2 molekul·ng −1 (RNA) untuk semua jaringan. Nilai rata-rata (semua log2) untuk ekspresi total ERV di tuba fallopi adalah 9,27, di ovar 7,2 dan di HGSOC 10,1; untuk L1-5′UTR di tuba fallopi 14,24, di ovar 12,94 dan HGSOC 15,88; untuk L1-ORF2 di tuba fallopi 16,07, di ovar 10,23 dan di HGSOC 14,79. Batang grafik menggambarkan SEM. Uji Mann–Whitney U dua sisi digunakan untuk nilai- P . ** P : 0,0058; *** P : 0,0007; **** P < 0,0001. (B) Peta panas HGSOC ( n = 102) yang mengarah pada penemuan lima klaster ERV-L1 Peradangan signifikan yang berbeda menggunakan perangkat lunak komputer jmp . Ekspresi gen dari klaster tumor HGSOC yang mengidentifikasi lima klaster peradangan ERV – L1 yang signifikan dalam konteks 99 tanda tangan gen pengatur Interferon (IRGS) menggunakan teknologi NanoString. Peradangan = INF; ERV = retrovirus endogen; L1 = L1-ORF2 . Jumlah tumor dan % dari kohort HGSOC disertakan: ID Warna untuk kotak ungu = INF-rendah ERV-L1 rendah ( n = 33; 32,3%); kotak hijau = INF-rendah ERV-L1 tinggi ( n = 38; 37,2%); kotak emas = Triple-I ( n = 6; 5,9%); kotak biru = INF-tinggi ERV-L1 rendah ( n = 11; 10,8%); kotak merah = INF-tinggi ERV-L1 tinggi ( n = 14; 13,7%). Kunci = Skor Z untuk tingkat ekspresi gen kiri atas. Semua gen ERV-L1 dan 22 gen yang menunjukkan klaster Triple-I dikurung di sebelah kanan peta panas; lihat juga Tabel S1B. (C) Lima klaster ekspresi peradangan ERV-L1 HGSOC mengidentifikasi berbagai kelangsungan hidup bebas kekambuhan pasien (RFS) yang signifikan yang terkait dengan IRGS tertentu. Kurva kelangsungan hidup Kaplan–Meier untuk RFS menggunakan perangkat lunak JMP ditunjukkan (Log rank P = 0,018). Bagian bawah menunjukkan jumlah pasien yang terstratifikasi yang berisiko dari 0 hingga 66 bulan. Kunci warna untuk setiap peradangan (INF), klaster ekspresi ERV-L1 (retrovirus endogen); L1 ( L1-ORF2 ) ditunjukkan di sebelah kanan. Warna cocok dengan peta panas dalam (B).
3.2 Lima kelompok peradangan HGSOC ERV-L1-ORF2 yang berbeda menentukan kelangsungan hidup
HGSOC diklasifikasikan terutama sebagai tumor ‘dingin’ atau tidak meradang dengan jumlah TIL yang berkurang, berbeda dengan tumor ‘panas’ atau meradang [ [ 45 , 46 ] ]. Namun, jumlah TIL yang lebih tinggi dapat ditemukan dalam sebagian kecil HGSOC, sehingga meningkatkan kelangsungan hidup pasien, yang mendukung aktivitas antitumor [ [ 32 ] ]. Tumor meradang yang diperkaya dengan sel T berasosiasi dengan tanda tangan gen peradangan, di mana beberapa tumor mengekspresikan ligan titik pemeriksaan imun PD-L1 secara berlebihan, penanda penghindaran imun tumor [ [ 45 ] ]. Kami pertama kali menguji apakah tingkat tinggi ERV dan ekspresi gen L1-ORF2 (ditandai sebagai L1 ) berkorelasi positif dengan peradangan tinggi dan kelangsungan hidup pasien dengan HGSOC yang lebih baik dan sebaliknya. Dengan menerapkan panel gen NanoString kami, kami memilih 99 gen (Tanda Tangan Gen Peradangan dan Pengatur = IRGS) yang berkorelasi dengan respons imun (Gbr. 1B , Tabel S1B ). IRGS mencakup sensor asam nukleat, anggota jalur IFN, sel imun, sitokin, dan regulator imun lainnya. Beberapa gen terpilih tumpang tindih dengan tanda tangan gen inflamasi yang dipublikasikan sebelumnya [ [ 45 ] ]. Dengan menggunakan analisis pengelompokan hierarkis tanpa pengawasan dan mengkorelasikan ekspresi gen ERV-L1 HGSOC ( n = 102) dengan IRGS, kami mengidentifikasi lima kluster tumor berbeda dengan kelangsungan hidup bebas kekambuhan pasien yang berbeda secara signifikan (RFS; 5,5 tahun) ( P : 0,018) (Gbr. 1B,C , Tabel 1 ). Seperti yang diprediksi, satu kluster menunjukkan inflamasi tinggi (inflamasi tinggi) dengan ekspresi ERV-L1 tinggi ( ERV-L1 tinggi) (13,7%), yang berkorelasi positif dengan kelangsungan hidup pasien yang lebih lama (Gbr. 1B,C , Tabel 1 ). Tanpa diduga, kluster tumor yang lebih kecil dengan inflamasi tinggi tetapi ekspresi gen ERV-L1 rendah (5,9%) secara signifikan menunjukkan kelangsungan hidup pasien terbaik. Kelompok tumor unik ini menunjukkan ekspresi gen tinggi dari 22 gen, termasuk pengatur titik pemeriksaan imun, seperti CTLA4 , TIGIT , LAG3 , dan CD27 , penanda permukaan sel imun, dan gen inflamasi (Gbr. 1B,C dan Tabel 1 , Tabel S1B ). Kami menyebut kelompok ini Triple-I, untuk sel Imun tinggi, Peradangan, dan titik pemeriksaan imun.
Sebaliknya, tiga kelompok tumor yang tersisa menunjukkan kelangsungan hidup pasien terburuk (Gbr. 1B,C dan Tabel 1 ). Satu kelompok tumor menandakan peradangan rendah dengan ekspresi ERV-L1 rendah (peradangan- ERV-L1 rendah rendah) (32,3%), mendukung kemungkinan represi elemen ERV , seperti metilasi dan aktivitas antitumor sel imun yang lebih rendah. Kelompok tumor terbesar secara tak terduga menunjukkan peradangan rendah dengan ekspresi gen ERV-L1 tinggi (peradangan- ERV-L1 rendah tinggi) (37,2%), menunjukkan blok yang memulai peradangan meskipun ekspresi ERV-L1 meningkat . Kelompok tumor terakhir dikaitkan dengan peradangan tinggi dan ekspresi ERV-L1 rendah (peradangan- ERV-L1 tinggi rendah) (10,8%). Semua analisis di atas menunjukkan sifat intrinsik dan kompleks HGSOC dalam konteks ekspresi gen ERV-L1 dan peradangan yang berkaitan dengan kelangsungan hidup pasien.
Setelah mengidentifikasi lima klaster peradangan ERV-L1 HGSOC signifikan yang berhubungan dengan kelangsungan hidup yang beragam, kami selanjutnya menguji klaster ini dengan variabel klinis dan patologis menggunakan analisis Regresi Cox (Tabel 1 , Tabel S1H,I ). Variabel-variabel berikut dianalisis dan disesuaikan secara multivariabel, seperti penerapan kemoterapi adjuvan, stadium pT, status reseksi (R), stadium pN (stadium limfonodal) dan invasi limfovaskular. Hasil penelitian menunjukkan bahwa hanya pT3 versus pT1, kemoterapi (ACT) dan fenotipe klaster tumor yang merupakan faktor independen signifikan untuk kelangsungan hidup (Tabel S1H ). Yang penting, kelompok Triple-I tetap sebagai prediktor independen dari RFS yang membaik jika dibandingkan dengan ERV-L1 rendah inflamasi (HR = 0,09; P = 0,002), ERV-L1 tinggi inflamasi (HR = 0,09; P = 0,002) dan ERV-L1 rendah inflamasi (HR = 0,14; P = 0,038) (Tabel S1H ). Selain itu, kelompok Triple-I juga tetap sebagai prediktor independen dari RFS yang membaik dengan menganalisis ulang regresi Cox yang disesuaikan multivariabel untuk kelompok Triple-I versus semua kelompok klaster lainnya bersama-sama (HR = 0,14; P = 0,0127) (Tabel S1I ).
Bahasa Indonesia: Untuk memperoleh wawasan tambahan ke dalam sifat molekuler dari lima kelompok tumor unik dengan kelangsungan hidup pasien, kami menganalisis secara lebih rinci setiap kelompok dalam hal ekspresi ERV-L1 dengan IRGS (Tabel S1B ). Analisis ini juga mencakup sel-sel imun yang dikuantifikasi menggunakan (a) CIBERSORT [ [ 43 ] ], yang menghitung sel-sel imun yang berbeda dari profil ekspresi gen; (b) perangkat lunak qupath untuk mengkuantifikasi sinyal hibridisasi ekspresi protein IHC; dan (c) H&E untuk menentukan persentase sTIL. Mengenai kelompok tumor ERV-L1 – tinggi peradangan-tinggi, hasil menunjukkan bahwa ekspresi tinggi ERV-L1 , kecuali HRES1 , dikaitkan dengan respons imun bawaan (Gbr. 2A,B ). Misalnya, induksi pensinyalan IFN tipe I klasik melalui dsRNA kemungkinan berasal dari transkrip ERV sense dan antisense [ [ 6 ] ], transkrip dwiarah L1 , atau hibrid RNA:DNA [ [ 21 , 23 ] ]. Hasil menunjukkan ekspresi tinggi dari (a) TLR3 sitosolik dan membran, 4, 9 ; (b) sensor untuk dsRNA, misalnya, MDA5 , RIG-I ; atau untuk dsDNA , misalnya, cGAS atau bentuk Z dari RNA atau DNA, misalnya, ZBP1 ; (3) ISG, seperti IFI16 , AIM2 , OASL ; faktor pengatur interferon ( IRF1-9 ); (d) faktor transkripsi NFKB1 dan NFKB2 ; (e) pengatur pembatas jalur ADAR1 , SAMHD1 ; (f) sel imun, pengatur titik pemeriksaan imun, dan kemokin ( CCL3 , CCL4 , dan CXCL8-11 ) dan (g) sTIL tinggi (Gbr. 2A ). Bersama dengan jumlah sedang sel imun CD4 + dan CD8 + , yang penting, makrofag M1 juga diperkaya dalam klaster ERV-L1 tinggi inflamasi , sedangkan makrofag M2 lebih rendah (Gbr. 3A–C ). Semua hal di atas sejalan dengan kelangsungan hidup pasien yang lebih baik (Gbr. 1C ).

Gambar 2
Buka di penampil gambar
Kekuatan Gambar
Peta panas menunjukkan perbedaan tingkat ekspresi ERV-L1 dan atau tanda tangan gen pengatur peradangan (IRGS) antara lima klaster tumor (lihat Tabel 1 untuk nomor tumor dan % yang mewakili klaster ERV-L1 peradangan , dan Tabel S1B untuk gen IRGS). Peradangan (INF); ERV = retrovirus endogen dan L1 ( L1-ORF2 ). (A) Peta panas IRGS dengan beberapa gen tambahan yang mewakili berbagai fungsi peradangan dengan lima klaster tumor. NanoString menghitung dalam log2 dari tingkat ekspresi gen yang berbeda. Klaster tumor: Dari Atas ke bawah: ChemoK = CCL3,4; CXCL8,9,10,11; NLRP1 ; NFKB = NFKB1 /2, TBK1 ; RNAse = RNAseH1, RNAseH2A, RNAseL, XRN1,2, DICER, DROSHA ; IRF-STAT = IRF1,2,3,5,7,8,9; STAT1,3 ; Sensor = cGAS (MB21D1); IFI16; TUJUAN2; DDX41; STAU1/2; OAS1; OASL; DHX9; STING1 (TMEM173); ZBP1 ; RLR = MAVS; RIG-I (DDX58); DHX58; IFIH1 ; Defensin = IFI27,35,44,44L; ISG15,20; SETDB1; KAT5 ; Pembatas = APOBEC3A,3B; ADAR1; SAMHD1; PANGKAS22,28,40; TREX1; IFI6; MOV10; MX1,2; ZC3HAV1; RIF1; IFITM1,3 ; ICP = titik pemeriksaan imun = LAG3, CTLA4, PDCD1LG2 (PD-L2), PDCD1 (PD-1), CD274 (PD-L1), CD27, HAVCR2 (TIM-3); TIGIT, VTCN1, NECTIN4 ; IL + TNFA = IL6,10,18,1B , TNFA ; TLR = TLR3,4,9 ; IFNE ; IFNG; Imun = sel imun CD3z (CD247); CD4; CD8A; CD44; CD68; CD80; CD163; FOXP3; GZMB; PRF1; NKG7; EOMES ; IFNA,B1 dan TIL = sTIL dalam % dianalisis oleh H&E untuk setiap klaster tumor: INF-tinggi ERV-L1 tinggi: 11,92%; INF-tinggi ERV-L1 rendah: 8,9%; Triple-I: 22,83%; INF-rendah ERV-L1 tinggi: 9,5%; INF-rendah ERV-L1 rendah: 3,3%. (B) Peta panas hasil PCR real-time kuantifikasi absolut untuk ERV tunggal dan tingkat ekspresi elemen ERV total (kanan bawah) untuk setiap klaster ERV-L1 inflamasi yang disebutkan di bawah ini. (C) CXCL9 dan AIM2induksi ekspresi gen setelah pengobatan IFNG pada lini sel karsinoma ovarium serosa tingkat tinggi (HGSOC). Sumbu Y = nilai ekspresi gen 2-DDcT (log2) dari CXCL9 dan AIM2 ; Sumbu X = pengobatan IFNG (21 jam) dari lini sel TyKnu dan Kuramochi HGSOC; kontrol yang tidak diobati (ctrl) (hijau), 10 ng·mL −1 , 50 ng·mL −1 IFNG (abu-abu), n = 6. Yang ditampilkan adalah kotak dan kumis dengan nilai minimum hingga maksimum. Signifikansi dihitung menggunakan uji Mann–Whitney U dua sisi . (D) Peta panas hitungan NanoString dalam log2 dari 26 tingkat ekspresi gen yang berbeda yang berkaitan dengan gen tunggal dari IRGS dengan penyorotan gen terkait piroptosis.
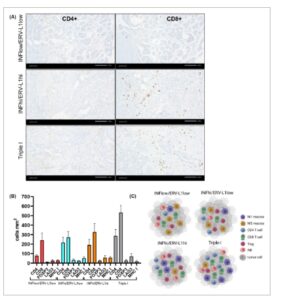
Gambar 3
Buka di penampil gambar
Kekuatan Gambar
Karsinoma ovarium serosa tingkat tinggi (HGSOC) peradangan klaster ekspresi gen ERV-L1 dan asosiasi dengan sel imun; Peradangan = INF, ERV = retrovirus endogen, L1 = L1-ORF2 . (A) Jaringan HGSOC dari klaster gen peradangan ERV-L1 (lihat Tabel 1 untuk klaster tumor) dihibridisasi dengan antibodi CD4 dan CD8 menggunakan IHC. Ini merupakan replikasi biologis independen dari tumor peradangan-rendah ERV-L1 rendah ( n = 33); peradangan-tinggi ERV-L1 rendah ( n = 11); peradangan-tinggi ERV-L1 tinggi ( n = 14) dan klaster tumor Triple-I ( n = 6 tumor). Foto mikroskopis IHC menunjukkan sel imun CD4 + dan CD8 + dari tiga klaster tumor yang mewakili peradangan-rendah ERV-L1 rendah (atas); peradangan-tinggi ERV-L1 tinggi (tengah) dan klaster tumor Triple-I (bawah). Batang di kanan bawah menunjukkan 100 μm. (B) Grafik yang menunjukkan kuantifikasi sel imun menggunakan perangkat lunak qpath (sel·mm −2 ) untuk klaster tumor tertentu (lihat Tabel 1 untuk jumlah tumor dan % dari setiap klaster tumor; n = X tumor yang dikuantifikasi per klaster tumor) peradangan-rendah ERV-L1 rendah ( n = 33 tumor dalam klaster ini); peradangan-tinggi ERV-L1 rendah ( n = 11 tumor dalam klaster ini); peradangan-tinggi ERV-L1 tinggi ( n = 14 tumor dalam klaster ini) dan klaster tumor Triple-I ( n = 6 tumor dalam klaster ini), yang juga merupakan contoh (A) dan (C). Batang grafik mewakili SEM. (C) Diagram skematik menunjukkan hierarki sel imun dari empat klaster tumor peradangan ERV-L1 yang representatif. Jumlah sel imun (%) dihitung menggunakan perangkat lunak cibersort , yang menerapkan banyak variabel ekspresi gen, dan dikonfirmasi sebagian menggunakan perangkat lunak q-path untuk kuantifikasi ekspresi IHC CD4 + , CD8 + dan Treg (FOXP3). Kuantifikasi CIBERSORT dari makrofag M1 (CD68) dan M2 (CD163), sel NK dan Treg di antara tuba Fallopi ( n = 13) = M1, M2 = 0%; NK = 1%; Treg = 4% (tidak ditampilkan di sini); dan empat kelompok tumor: peradangan-rendah /ERV/L1 -rendah ( n = 33): M1 = 5%; M2 = 12%; NK = 7%, Treg = 12%; peradangan-tinggi /ERV/L1 -rendah ( n = 11): M1 = 25%; M2 = 10%; NK = 5%, Treg = 18%; peradangan-tinggi /ERV/L1- tinggi ( n = 14): M1 = 8%; M2 = 2%; NK = 8%, Treg = 17%; Triple-I ( n = 6): M1 = 8%; M2 = 4%; NK = 11%, Treg = 7% (lihat Tabel 1 untuk jumlah tumor dan % dari setiap gugus tumor; n = X tumor yang dihitung per gugus tumor).
Klaster Triple-I dengan kelangsungan hidup pasien terbaik (Gbr. 1B,C ) mendukung peran inflamasi yang dimediasi IFNG. IFNG yang tinggi mengarah ke STAT dan ekspresi gen target yang lebih tinggi, misalnya, AIM2 , CXCL9 , dan IRF dan sebagai tambahan, ekspresi IFNA atau B1 yang rendah (Gbr. 2A ). Inflamasi tinggi dalam konteks dengan ekspresi ERV yang rendah merupakan hal baru karena tidak adanya mimikri virus (Gbr. 2B ). Selain itu jumlah sTIL tertinggi (22,83%) dalam klaster Triple-I diperkuat oleh ekspresi tertinggi penanda permukaan sel imun (Gbr. 2A dan 3A–C ). Yang penting, kami membuktikan dengan dua lini sel HGSOC TyKnu dan Kuramochi bahwa setelah penambahan IFNG baik AIM2 dan CXCL9 meningkat secara signifikan (Gbr. 2C ). Temuan ini menunjukkan bahwa sel HGSOC merupakan salah satu sumber tingkat ekspresi gen AIM2 dan CXCL9 yang tinggi yang ditemukan dalam klaster Triple-I, di mana kedua gen tersebut merupakan target IFNG. Untuk tumor Triple-I, selain gen pembunuh tumor sel T sitotoksik yang dimediasi IFNG, seperti GZMB, ekspresi AIM2 yang tinggi bersama dengan ekspresi NLRP1 mendukung peran piroptosis melalui inflamasom (Gbr. 2D ).
Kami kemudian menilai kohort HGSOC kami menggunakan variabel spesifik lain yang berhubungan dengan kelangsungan hidup pasien untuk menguatkan temuan kami di atas dari kedua klaster tumor ERV-L1 tinggi-inflamasi dan Triple-I. Kelangsungan hidup pasien yang jauh lebih lama berhubungan dengan jumlah sTIL yang lebih tinggi ( P : 0,037) dan sel T CD4 + ( P : 0,0365) (Gbr. 4A,B ), yang didukung oleh literatur [ [ 47 ] ]. Kelangsungan hidup yang lebih baik berkorelasi secara signifikan dengan ekspresi gen AIM2 yang lebih tinggi ( P : 0,0066, Gbr. 4C ) dan CXCL9 ( P < 0,0001, Gbr. 2D dan 4D ). Dengan menggunakan uji statistik ‘pemangkasan’, ekspresi CXCL9 yang tinggi mengidentifikasi klaster Triple-I dengan presisi 100%. Untuk menguji signifikansi klaster Triple-I, kami menganalisis kohort HGSOC dari TCGA ( n = 294) menggunakan panel 22 gen dengan ekspresi berlebih tertinggi yang spesifik untuk klaster ini (Gbr. 1B,C ; Tabel S1B ). Hebatnya, hasil menunjukkan bahwa CXCL9 merupakan kontributor utama untuk kelangsungan hidup, khususnya klaster Triple-I HGSOC ( P : 0,0298, Gbr. 4F ), sedangkan AIM2 tidak menunjukkan signifikansi untuk kohort TCGA.
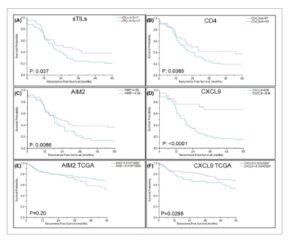
Gambar 4
Buka di penampil gambar
Kekuatan Gambar
Landmark TIL dan gen untuk kelangsungan hidup pasien bebas kekambuhan (RFS) yang signifikan dari karsinoma ovarium serosa tingkat tinggi (HGSOC) ( n = 102) dan analisis TCGA dengan HGSOC ( n = 294). Kurva kelangsungan hidup Kaplan–Meier untuk RFS menggunakan perangkat lunak komputer jmp dengan kohort HGSOC kami. (A–D) dan TCGA (E, F); (A) sTIL; (B) CD4 + ; (C) AIM2 ; (D) CXCL9 ; (E) TCGA ( n = 294) AIM2 ; (F) TCGA ( n = 294) CXCL9 .
Berbeda dengan yang di atas, mengenai klaster tumor ERV-L1 rendah peradangan ‘dingin’ , semua gen ERV-L1 dan IRGS tidak menunjukkan induksi, kecuali ERV tunggal HRES1 yang diinduksi ke tingkat yang lebih tinggi (Gbr. 2A,B,D ). Sebaliknya, klaster tumor rendah peradangan ‘dingin’ terbesar dikaitkan dengan ERV tinggi (tinggi ERV-L1 rendah peradangan ; 37,2%). Meskipun tingkat ekspresi ERV tinggi, yang sebanding dengan klaster tinggi ERV-L1 tinggi peradangan , kami memeriksa kemungkinan ekspresi sensor dsRNA yang abnormal (Gbr. 2A,B ; Gbr. S3 ). Memang, ekspresi gen dari kedua sensor dsRNA MDA5 dan RIG-I secara signifikan lebih rendah dalam klaster tumor tinggi ERV-L1 rendah peradangan , yang mendukung blok abnormal dalam pensinyalan IFN (Gbr. S3 ). Yang penting, ekspresi sensor dsRNA yang rendah sebanding dengan tumor ERV-L1 rendah akibat peradangan dan Triple-I, yang menunjukkan tidak adanya pensinyalan karena ERV dan dsRNA L1 . Namun, hal ini berbeda dengan klaster ERV-L1 tinggi akibat peradangan, di mana reseptor MDA5 dan RIG-I tinggi dan menunjukkan respons IFN.
Mengingat ekspresi gen ERV-L1 yang rendah dalam klaster tumor ERV-L1 rendah ‘panas’ yang lebih kecil (10% dari semua HGSOC) dengan kelangsungan hidup RSF pasien yang buruk, kami mencatat kadar MDA5 dan RIG-I yang tinggi (Gbr. S3 ). Meskipun kami tidak memeriksa elemen-elemen yang tersebar pendek (SINE) seperti Alu RNA, yang mengikat RIG-I yang menginduksi peradangan, data kami dapat mendukung kemungkinan peran Alu RNA atau elemen berulang lainnya yang memediasi peradangan mengenai klaster ini. Lebih jauh, kami menentukan perbedaan antara ERV-L1 rendah yang tinggi peradangan dan klaster Triple-I, yang dapat membantu menjelaskan kelangsungan hidup pasien yang berlawanan (Gbr. 1C ). Hasil penelitian mengungkapkan bahwa: (a) kelompok Triple-I memiliki sTIL yang lebih tinggi (22,8% vs. 8,9%) dan sel NK (11% vs. 5%) jika dibandingkan dengan klaster ERV-L1 rendah yang tinggi peradangan, masing-masing; (b) klaster ERV-L1 rendah yang berinflammasi tinggi menunjukkan kandungan sel Treg inhibitor yang lebih tinggi daripada kelompok Triple-I (18% vs. 7%), masing-masing (Gbr. 3B,C ; CIBERSORT); dan (c) kelompok Triple-I menunjukkan tingkat ekspresi gen titik pemeriksaan imun VTCN1 (disebut juga B7-H4 ) yang 15,7 kali lebih rendah jika dibandingkan dengan klaster tumor ERV-L1 rendah yang berinflammasi tinggi (Gbr. 2D ).
Terakhir, untuk menentukan tanda tangan global ERV-L1 minimal yang menentukan guna membedakan antara ERV-L1 tinggi dan ERV-L1 rendah dengan peradangan tinggi atau rendah, kami memilih ERV dengan ekspresi diferensial tertinggi (Gbr. 2B ). Hasil penelitian menunjukkan bahwa tanda tangan ekspresi ERV3 , ERV-Kenv/pol , ERV-H , dan ERV-Wgag , serta L1 sudah mencukupi.
3.3 Reseptor titik pemeriksaan imun dan interaksi ligan baru dalam kelompok tumor
Reseptor kematian terprogram 1 (PD-1) diekspresikan pada sel T setelah penyajian antigen dan aktivasi sel T untuk melemahkan dan mengendalikan respons. Regulasi penghambatan terjadi setelah pengikatan PD-1 ke ligan PD-L1 atau PD-L2, yang diekspresikan pada sel penyaji antigen (APC) atau dengan sel tumor yang menyebabkan penghindaran [ [ 48 ] ]. Untuk menentukan peran protein PD-1 dan PD-L1 dalam HGSOC TIME, kami mengukur ekspresi protein mereka. Untuk kelima klaster HGSOC, ekspresi protein PD-1 dan PD-L1 secara eksklusif berasal dari sel imun, tanpa ekspresi protein PD-L1 yang terdeteksi pada sel tumor (Gbr. S4A–C ). Hasil ini mendukung tidak adanya adaptasi PD-L1 tumor yang menyebabkan kelelahan sel T dalam TIME. Baik klaster tumor Triple-I maupun ERV-L1 tinggi Peradangan menunjukkan tingkat sel imun tertinggi dengan ekspresi protein PD-1 dan PD-L1 di garis depan invasi, sedangkan semua klaster tumor lainnya diekspresikan rendah untuk kedua titik pemeriksaan. Kami beralasan bahwa ekspresi membran PD-1 yang lebih tinggi pada sel T bersama dengan peningkatan kadar APC PD-L1 mencerminkan homeostasis sel imun normal, menghasilkan populasi sel T dengan respons antitumor yang meningkat, sehingga berkontribusi pada kelangsungan hidup pasien yang lebih lama (Gbr. 1C ). Ekspresi PD-L1 yang tinggi pada sel imun yang berhubungan dengan kelangsungan hidup pasien yang lebih baik didukung oleh literatur [ [ 41 , 47 ] ].
Reseptor CD28 diekspresikan secara konstitutif pada sel T dan, setelah presentasi antigen, menghasilkan keadaan aktif penuh yang mengarah pada ekspansi klonal, sitokin, dan sekresi enzim yang diperlukan untuk menargetkan sel yang terinfeksi virus atau tumor [ [ 49 ] ]. Untuk mengatur aktivasi sel T secara negatif, protein terkait limfosit T sitotoksik 4 (reseptor CTLA4) diekspresikan bersama dengan CD28 di membran. Karena homologi asam amino yang serupa, kedua reseptor bersaing untuk ligan CD80 yang sama pada APC [ [ 49 ] ]. Atenuasi sel T ini terjadi pada tahap awal aktivasi sel T setelah presentasi antigen, misalnya, di kelenjar getah bening. Ekspresi gen yang tinggi dari reseptor CTLA4 merupakan karakteristik utama dari klaster tumor Triple-I (Gbr. 1C dan 2A , Gbr. S5 ). Bahasa Indonesia: Untuk membedakan lokalisasi sitosolik atau membran CTLA4, bersama dengan rasionya terhadap CD28, kami mengukur dan membandingkan kadar protein CTLA4, CD28, dan ligan CD80 di kelenjar getah bening dan di TIME antara pasien yang sama yang mewakili gugus tumor tertentu (Gbr. S5A–E ). Membandingkan jaringan kelenjar getah bening menunjukkan bahwa CD28 adalah titik pemeriksaan yang diekspresikan paling signifikan di kelenjar getah bening dibandingkan dengan CD80 dan CTLA4 membran, yang memiliki jumlah yang sama menggunakan IHC (Gbr. S5A,D,E ). Hasil ini mendukung aktivasi sel T setelah presentasi antigen yang terjadi di kelenjar getah bening untuk ketiga gugus tumor. Namun, membandingkan jaringan tumor yang cocok dengan pasien menunjukkan CD28 adalah yang tertinggi yang diekspresikan dalam TIME untuk gugus Triple-I dibandingkan dengan gugus tumor lainnya (Gbr. S5B ). Korelasi ekspresi protein dikonfirmasi dalam TIME di antara kelima gugus tumor HGSOC ( n = 102) (Gbr. 5A–C ). Karena CD28 merupakan status sel T yang aktif, kami melakukan IHC multipleks untuk mengetahui rasio ko-ekspresi CD28:CTLA4 pada sel imun (Gbr. 5D–G , Gbr. S5C ). Hasil penelitian menunjukkan bahwa kedua klaster dengan inflamasi tertinggi (Triple-I dan Inflamasi-tinggi ERV-L1 tinggi) menunjukkan populasi sel imun yang besar dalam TIME, yang hanya mengekspresikan CD28 tanpa ekspresi protein CTLA4. Hasil ini mendukung aktivasi sel T, di mana CD28 mengikat CD80 tanpa kompetisi penghambatan yang dimediasi oleh CTLA4. Sejalan dengan kelangsungan hidup pasien terbaik (Gbr. 1C ), kami mengusulkan bahwa sebagai respons terhadap antigen tumor dalam kelenjar getah bening, pengatur titik pemeriksaan pengaktif CD28, serta kemungkinan yang lain, mewakili status sel T yang lebih aktif dalam TIME HGSOC, terutama yang berkaitan dengan klaster Triple-I (Gbr. 5F ).
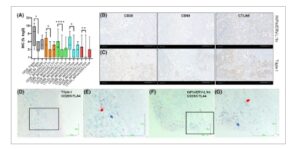
Gambar 5
Buka di penampil gambar
Kekuatan Gambar
Perbandingan ekspresi protein pengatur titik pemeriksaan imun (CD28, CD80, dan CTLA4) antara lima klaster ERV-L1 inflamasi pada karsinoma ovarium serosa tingkat tinggi (HGSOC) ( n = 102 jaringan) menggunakan IHC. (A) Grafik menggambarkan IHC yang dikuantifikasi menggunakan perangkat lunak q-path dari CD28, CD80, dan CTLA4 (% sel log2) dengan perbandingan kelima klaster tumor ERV-L1 inflamasi dengan perbedaan signifikan menurut uji Mann–Whitney U dua sisi : Triple-I ( n = 6 replikasi independen biologis) CD28 vs. CD80: * P : 0,0476; ERV-L1 tinggi inflamasi tinggi ( n = 14 replikasi independen biologis) CD28 vs. CD80: * P : 0,0103; peradangan-rendah ERV-L1 tinggi ( n = 38 replikasi independen secara biologis) CD28 vs. CD80: **** P < 0,0001; peradangan-tinggi ERV-L1 rendah ( n = 11 replikasi independen secara biologis) CD28 vs. CD80: * P : 0,0281; peradangan-rendah ERV-L1 rendah ( n = 33 replikasi independen secara biologis) CD28 vs. CD80: ** P : 0,0049. Yang ditampilkan adalah kotak dan kumis dengan nilai minimum hingga maksimum. (B, C) Foto representatif dari dua kelompok tumor (peradangan-tinggi ERV-L1 tinggi dan Triple-I) menunjukkan ekspresi IHC dari CD28, CD80 dan CTLA4. (D–G) Multiplex IHC menggunakan dua deteksi warna CD28 dan CTLA4 dilakukan dengan jaringan tumor primer dari klaster Triple-I ( n = 6 replikasi independen secara biologis) dan klaster ERV-L1 tinggi inflamasi ( n = 3 replikasi independen secara biologis). Foto-foto menunjukkan contoh CD28 (warna hijau/biru) dan CTLA4 (merah) dari jaringan primer dari tumor Triple-I dan ERV-L1 tinggi inflamasi. Kotak dalam (D) dan (F) mewakili perbesaran (E) dan (G). Semua batang skala ditunjukkan di kanan bawah sebagai 100 μm. Lihat Gambar 5G untuk kuantifikasi ekspresi protein CD28 dan CTLA4.
4 Diskusi
Sejak penemuan mimikri virus melalui ERV yang memicu respons IFN tipe I dalam berbagai tumor, termasuk HGSOC, banyak pengetahuan mengenai pentingnya retroelemen dan imunitas bawaan telah ditunjukkan [ [ 6 , 50 ] ]. Reaktivasi ekspresi elemen ERV yang menghasilkan tingkat dsRNA yang lebih tinggi dalam sel tumor adalah jalur kanonik yang memicu peradangan dan memprediksi kelangsungan hidup pasien yang lebih baik [ [ 6 , 7 , 29 , 51 , 52 ] ]. Studi kami saat ini membantu untuk menentukan tingkat ekspresi gen ERV dan L1 dan kontribusinya terhadap peradangan dalam WAKTU HGSOC yang penting untuk kelangsungan hidup pasien menggunakan jaringan tumor sebelum pengobatan. Pemeriksaan 102 HGSOC dalam konteks dengan ekspresi ERV-L1 dan peradangan menunjukkan sebagian besar tumor ‘dingin’ atau tidak meradang (69,5%), tetapi menariknya terkait dengan ekspresi ERV-L1 rendah atau ekspresi ERV-L1 tinggi yang dikaitkan dengan kelangsungan hidup yang lebih buruk. Tumor ‘dingin’ ini mengungkapkan ekspresi IRGS yang lebih rendah secara signifikan, terutama menyoroti pentingnya sensor dsRNA MDA5 dan RIG-I , sTIL yang lebih rendah, misalnya, CD4 + , dan asosiasi yang lebih kuat dengan makrofag M2. Bahkan meskipun ekspresi ERV-L1 tinggi tercatat dalam klaster tumor ERV-L1 rendah peradangan ‘dingin’ , kami menemukan kadar rendah MDA5 dan RIG-I yang tidak normal yang mendukung respons tumor adaptif dalam TIME karena blok yang memicu peradangan. Temuan terbaru dari literatur juga menunjukkan gen deregulasi lain yang terlibat dalam pensinyalan IFN, yang berkontribusi pada respons adaptif tumor. Misalnya, helicase DHX9 yang diidentifikasi sebagai represor untuk dsRNA dan dsDNA yang disebabkan oleh kerusakan DNA diekspresikan secara berlebihan pada kanker paru-paru sel kecil [ [ 53 ] ]. Namun, kami tidak menemukan perbedaan ekspresi DHX9 yang signifikan antara kelima klaster tumor dalam kohort HGSOC kami.
Penemuan dua klaster tumor meradang ‘panas’ yang berbeda (total 19,6%) dalam kohort HGSOC kami dengan kelangsungan hidup pasien terbaik, tetapi dengan ekspresi ERV-L1 tinggi atau rendah yang berlawanan adalah hal baru dan mendukung pensinyalan IFN yang berbeda. Misalnya, sangat mencolok bahwa klaster tinggi ERV-L1 peradangan yang mewakili 13,7% HGSOC menunjukkan sensor dsRNA, IFNA , IFNB1 , dan pensinyalan IFNG yang paling ditingkatkan . Bersama-sama jalur ini menghasilkan kadar ISG, sitokin, dan sTIL yang tinggi dan mendukung respons imun bawaan. Di sisi lain, kami mengusulkan bahwa IFNG memainkan peran yang lebih mengatur dalam klaster Triple-I dengan jumlah sTIL dan sel T CD4 + tertinggi dan ekspresi ERV-L1 terendah , yang mendukung respons imun adaptif. Hasil-hasil ini berbicara tentang peran langsung pembunuhan sel tumor oleh sel T, yang kami dukung disebabkan oleh peningkatan antigen tumor dan ekspansi sel T yang berasal dari kelenjar getah bening. Dukungan lebih lanjut untuk sel T sitotoksik yang sangat aktif berasal dari temuan kami bahwa CD28 hanya diekspresikan pada persentase tinggi sel imun tanpa regulator negatif CTLA4 dalam TIME. Sel T CD4 + sitotoksik telah diidentifikasi dalam kanker dan dikaitkan dengan ekspresi GZMB dan Perforin (PRF) melalui faktor transkripsi EOMES [ [ 54 ] ]. Yang penting, kami menemukan EOMES sangat teregulasi dalam klaster ERV-L1 -tinggi peradangan tinggi dan GZMB dan PRF dengan klaster tumor Triple-I (Gbr. 1B dan 2A ).
Sejalan dengan hal di atas, khususnya CXCL9 diekspresikan secara tinggi baik dalam klaster ERV-L1 -tinggi inflamasi maupun klaster Triple-I. Namun, jelas bahwa ekspresi CXCL9 tertinggi secara signifikan mewakili kontributor utama untuk kelangsungan hidup terbaik dalam kelompok Triple-I serta dengan analisis TCGA. Satu penjelasannya adalah bahwa sitokin ini menarik sel T yang diaktifkan dan memori, sehingga menekan pertumbuhan tumor [ [ 55 ] ]. Sebelumnya, telah ditunjukkan bahwa ekspresi protein CXCL9 dan CXCL10 yang tinggi meningkatkan kelangsungan hidup pasien HGSOC dan berasosiasi positif dengan TIL [ [ 56 ] ]. Studi ini juga menunjukkan bahwa sekresi protein CXCL9 terjadi dari lini sel kanker ovarium setelah penambahan IFNG dan TNFA menggunakan kultur sel, yang dibuktikan oleh temuan kami bahwa CXCL9 serta AIM2 adalah gen target untuk IFNG. TIL dan makrofag M1 mengeluarkan CXCL9 [ [ 57 ] ], tetapi temuan di atas mendukung bahwa sel tumor, terutama dari klaster Triple-I, juga mengeluarkan CXCL9 dalam TIME, yang mengarah pada perekrutan TIL lebih lanjut. Skenario ini dapat menjelaskan temuan kami tentang sTIL dan makrofag M1 tertinggi yang terkait dengan klaster tumor Triple-I.
Pengapian jalur IFN juga penting untuk memulai kerusakan DNA dan apoptosis yang mengakibatkan kematian sel tumor dalam TIME. Selain apoptosis, piroptosis menggambarkan kematian sel inflamasi dengan pembentukan inflammasom, pecahnya membran sel dengan dsDNA, dan pelepasan IL18 dan IL1B [ [ 4 ] ]. Gen utama yang terlibat dalam inflammasom adalah AIM2 , GSDM , NLR , dan Kaspase . Jaringan pensinyalan ini mengisyaratkan regulasi yang bermanfaat dari sensor, inflamasi, dan apoptosis atau piroptosis. Sebuah studi yang menganalisis gen piroptosis dari kohort pasien kanker ovarium TCGA menunjukkan bahwa AIM2 meningkat pada tumor ovarium dan berkontribusi pada kelangsungan hidup keseluruhan yang lebih lama [ [ 58 ] ]. Kami menemukan dukungan untuk inflammasom pada tumor Triple-I karena temuan kami bahwa AIM2 dan NLRP1 adalah gen yang diekspresikan tertinggi dibandingkan dengan semua kluster (Gbr. 2D ). Selain AIM2 dan NLRP1, cGAS dan IFI16 juga mengikat dsDNA [ [ 59 , 60 ] ]. Sensor dsDNA terakhir adalah ISG, kecuali NLRP1, di mana IFI16 dan AIM2 diinduksi melalui IFNG [ [ 61 ] ], sebagaimana dikuatkan dengan studi kultur sel HGSOC kami. Berbeda dengan klaster ERV-L1 tinggi inflamasi, klaster Triple-I menunjukkan ekspresi IFI16 yang lebih rendah , yang dapat menambah inflammasome, karena protein IFI16B, yang menghambat pengikatan AIM2 ke dsDNA dan melemahkan inflammasome aktif [ [ 62 ] ]. Mengenai piroptosis dan anggota pensinyalan IFN kontributor, perlu dicatat bahwa profil ekspresi ERV-L1 tinggi inflamasi dan klaster Triple-I sebagian kontras. Misalnya, IDO1 , AIM2 , CXCL9 , NLRP1 , dan GZMB menunjukkan ekspresi tertinggi dalam klaster Triple-I, sedangkan IL8 , IL6 , GSDMC , IFNA , dan IFNB1 menonjol dalam klaster ERV-L1 – tinggi peradangan tinggi (Gbr. 2D ).
Terakhir, hasil kami menunjukkan bahwa hierarki overekspresi dan korelasi signifikan L1-5′UTR , L1-ORF2 , dan ERV dalam HGSOC akibat hipometilasi DNA menunjukkan signifikansinya dalam entitas kanker ini. Penting bahwa penelitian terbaru menemukan bahwa 53% pasien kanker memiliki retrotransposisi somatik akibat L1 yang diaktifkan kembali [ [ 63 ] ]. Untuk kanker ovarium, reaktivasi L1 menyebabkan mutasi dan heterogenitas genom dalam lini sel kanker ovarium [ [ 64 ] ]. Meskipun penelitian lebih lanjut diperlukan untuk mengungkap kompleksitas kontribusi retroelemen L1 dalam peradangan kanker, khususnya untuk L1-ORF2, kami mendukung gagasan bahwa mereka mewakili sumber dsRNA dan RNA: DNA yang dikaitkan dengan respons IFN. Selain itu, pol ERV (khususnya ERV-Kpols) juga harus dianggap sebagai asal hibrida RNA:DNA [ [ 65 ] ], yang harus dianalisis lebih lanjut untuk perannya dalam pensinyalan IFN.
5 Kesimpulan
Kami mengusulkan model untuk HGSOC, di mana ekspresi ERV-L1 tinggi dan ERV-L1 rendah dapat dicontohkan sebagai dua jalur peradangan yang berbeda, dengan dan tanpa mimikri virus (Gbr. 6A ). Mengorelasikan ekspresi total ERV dan L1 dengan gen IRGS yang diekspresikan secara diferensial paling signifikan memungkinkan perbandingan langsung dan menghasilkan skor peradangan. Analisis di atas menunjukkan bahwa bahkan ketika elemen ERV-L1 diekspresikan secara tinggi, skor peradangan bisa tinggi atau rendah dan sebaliknya. Studi kami menunjukkan kompleksitas molekuler dari HGSOC TIME, di mana tingkat ekspresi gen ERV-L1 berasosiasi dengan pensinyalan IFN, peradangan, regulasi protein titik pemeriksaan, dan aktivitas antitumor melalui TIL yang penting untuk kelangsungan hidup pasien. Penting untuk dicatat bahwa kohort HGSOC kami yang terdiri dari 102 pasien berukuran sedang, yang terdiri dari semua orang Eropa dengan kebangsaan dari Jerman ( n = 96), tetapi juga dari Polandia, Turki, dan Serbia ( n = 6). Oleh karena itu, penting untuk memperluas jumlah pasien, termasuk berbagai kebangsaan, guna memverifikasi setiap kelompok tumor, khususnya Triple-I dengan kelangsungan hidup pasien terbaik. Lebih jauh, melakukan RNA-seq jarak jauh (lebih dari 300 nt) dengan beberapa pembacaan untuk identifikasi retroelemen dan gen inflamasi yang lebih spesifik akan membantu memahami jalur regulasi. Menentukan metilasi DNA dan histon juga akan membantu lebih jauh untuk menyimpulkan regulasi epigenetik retroelemen. Secara keseluruhan, kami menunjukkan bahwa HGSOC terdiri dari kelompok-kelompok unik, khususnya satu kelompok inflamasi yang tidak memiliki mimikri virus yang terkait dengan kelangsungan hidup yang lebih lama. Kami mengusulkan agar kelompok-kelompok tumor unik ini dapat direpresentasikan di antara jenis-jenis tumor lainnya.
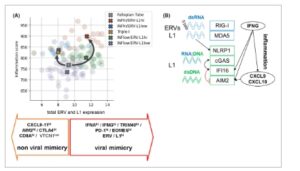
Gambar 6
Buka di penampil gambar
Kekuatan Gambar
(A) Model yang menunjukkan mimikri virus dan ‘nonvirus’ yang mengatur peradangan. Total molekul ERV-L1 (sumbu X ) dan skor peradangan ( sumbu Y ) untuk masing-masing dari lima klaster karsinoma ovarium serosa tingkat tinggi (HGSOC) dan tuba fallopi. Silakan lihat Bagian 2 untuk semua penjelasan statistik dan Tabel 1 untuk jumlah dan % klaster tumor dan tuba fallopi tertentu ( n = 13) yang diplot dalam grafik. Skor peradangan dihitung untuk setiap sampel tumor dari klaster peradangan rendah dan peradangan tinggi sebagai jumlah tingkat ekspresi log2 dari 99 gen IRGS. Lingkaran berwarna mewakili tumor tunggal dalam klaster tumor ERV-L1 peradangannya (cocok dengan ‘X’ berwarna dalam kotak inlay). Skor peradangan merangkum status peradangan HGSOC dalam metrik kontinu tunggal. Skor ini secara langsung dibandingkan dengan nilai ekspresi total ERV-L1 untuk setiap klaster ERV peradangan . Di bawah dalam kotak berbentuk panah adalah DEG paling signifikan untuk mimikri nonviral dan viral berdasarkan analisis dengan perubahan ekspresi minimal 4 kali lipat dan P yang disesuaikan < 0,01. Sebaliknya, VTCN1 , terutama yang berkaitan dengan klaster tumor Triple-I, mengalami penurunan regulasi dalam mimikri nonviral. Pemangkasan fitur digunakan untuk mendapatkan set gen terkecil yang terkait dengan klaster yang mewakili presisi tertinggi. Untuk mengidentifikasi tumor Triple-I dengan presisi 100%, hanya CXCL9 yang diperlukan. (B) Model menunjukkan regulasi molekuler untuk spesies asam nukleat yang berbeda (dsRNA, RNA:DNA, dsDNA) yang berasal dari ERV dan L1 dan dikenali oleh sensornya yang menyebabkan peradangan. Selain itu, kami dan yang lainnya menunjukkan bahwa sitokin peradangan IFNG menginduksi AIM2 dan pengatur negatifnya IFI16 , serta penanda peradangan lainnya seperti CXCL9 dan CXCL10.
